L'entrée d'une nouvelle séquence
La fenêtre d'entrée de séquence s'obtient par le menu Fichier/Entrée d'une séquence.
Prenons l'exemple d'entrée d'une séquence copiée dans la base de données NCBI :
Première étape : obtenir une ou des séquence à partir de la banque de données :
(cette étape peut être passée si on connaît déjà la séquence)
| A droite, on peut faire un tri parmi les organismes | 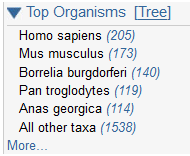 |
 |
Dans l'exemple ci-dessous, après avoir sélectionné Homo sapiens, les séquences 2 (partie codante : cDNA avec le codon stop)

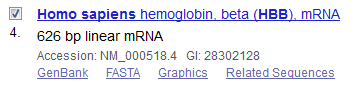
et 4 (ARNm) ont été sélectionnées.
-
Aller sur le site NCBI : http://www.ncbi.nlm.nih.gov/ et choisir la base de données (Database) « Nucleotid » pour ADN ou ARN ou « Protein » suivant ce que l'on cherche.
-
Taper le nom du gène ou de la protéine à rechercher : par exemple HBB pour la chaine β de l'hémoglobine.
-
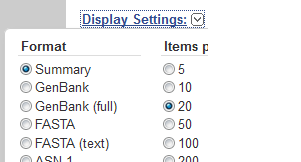
Choisir l'affichage (Display Settings) de type fasta (text) et valider avec le bouton Apply.
-
Sélectionner le texte et faire un clic droit pour faire apparaître le menu où l'on choisit Copier.
-
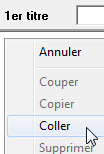
Dans GenieGen, après avoir choisi le menu Fichier/Entrée d'une séquence, Faire un clic droit sur la zone de texte et choisir Coller.
-
Attention les séquences ne seront correctement entrées que si la ligne de titre de chaque séquence (celle qui commence par >) tient bien sur une ligne. Ne pas y introduire de retour à la ligne, sinon ce qui est sur la deuxième ligne est considéré comme faisant partie de la séquence (en éliminant les caractères non conformes au codage).
Un aménagement utile est de mettre le nom de l'espèce entre crochets dans la ligne d'en-tête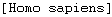 . Le logiciel l'utilisera alors comme nom d'espèce. Il est bon de conserver le code figurant en début d'en-tête
. Le logiciel l'utilisera alors comme nom d'espèce. Il est bon de conserver le code figurant en début d'en-tête 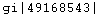 le logiciel le conservera dans le fichier edi enregistré. On aura alors un accès à la page correspondante de la base de données NCBI.
le logiciel le conservera dans le fichier edi enregistré. On aura alors un accès à la page correspondante de la base de données NCBI. -
Vérifier aussi que c'est le bon type de séquence qui est coché
 (ici ADN5'3') puis cliquer sur le bouton OK.
(ici ADN5'3') puis cliquer sur le bouton OK. -
Une fenêtre de vérification apparaît. Choisir l'onglet Données
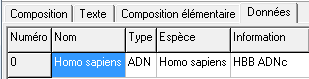 pour obtenir un tableau où l'on peut modifier le nom proposé pour chaque séquence ainsi que les autres colonnes. Valider pour entrer définitivement la ou les séquences.
pour obtenir un tableau où l'on peut modifier le nom proposé pour chaque séquence ainsi que les autres colonnes. Valider pour entrer définitivement la ou les séquences. -
Le tableau sera par la suite consultable avec le menu Informations de la fenêtre des séquences. La fiche NCBI sera consultable (s'il y a une liaison internet active) avec le menu
 . La fiche qui s'ouvrira dans le navigateur sera celle qui correspond à la ligne du tableau qui est sélectionnée. Il faut pour cela que le code NCBI de la séquence ait été enregistré.
. La fiche qui s'ouvrira dans le navigateur sera celle qui correspond à la ligne du tableau qui est sélectionnée. Il faut pour cela que le code NCBI de la séquence ait été enregistré. -
L'enregistrement des séquences se fait avec le menu Fichier/Enregistrer sous... cet enregistrement ne concerne que les séquences sélectionnées. Les fichiers enregistrés au format .edi (format Anagène) sont compatibles avec toutes les versions actuelles d'Anagène.
