Les relations entre les allèles d'un même gène (dominance/récessivité)
Par sbergeot
—
Dernière modification
08/04/2019 12:10
Les relations entre les allèles d'un même gène (dominance/récessivité)
Objectif : déterminer les génotypes de plusieurs individus ayant le même phénotype clinique.
Méthode :
- comparer les allèles que possède un individu avec les allèles de référence du gène xpc (xpc_adn.edi).
- en déduire son génotype.
- comparer les génotypes des individus de même phénotype clinique.
- proposer une explication à l'existence d'un descendant malade.
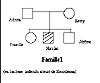
| Arbres généalogiques | Séquences des allèles des individus | Génotypes des individus |
| Famille 1 | allfam1.edi | Pour la famille 1
|
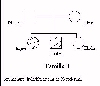
Famille 2 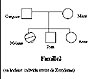 Cliquer pour agrandir |
allfam2.edi | Pour la famille 2
|
| Famille 3 | allfam3.edi | Pour la famille 3
|
Conclusion :
Un même phénotype peut correspondre à plusieurs génotypes :
- exemple : les génotypes (xpc_0/xpc_0) et (xpc_1/xpc_0) correspondent à un phénotype clinique "normal" (pas de Xeroderma).
- exemple : les génotypes (xpc_1/xpc_1) et (xpc_1/xpc_2) correspondent à un phénotype clinique "malade" (atteinte par le Xéroderma).
Les relations de dominance/récessivité entre les allèles peuvent expliquer que plusieurs génotypes peuvent conduire au même phénotype clinique : les allèles xpc_1 et xpc_2 sont récessifs par rapport à l'allèle xpc_0.