Activité pédagogique sur l’action des antiviraux contre le SARS-CoV-2
L’action des antiviraux contre le SARS-CoV-2
Problématique : Comment agissent les antiviraux ? A partir de l’analyse documentaire suivante vous allez déterminer comment des antiviraux ont pu être identifiés contre le coronavirus et expliquer leur mode d’action pour lutter contre l’infection virale.
Document 1 : étude du cycle du coronavirus SARS-CoV-2
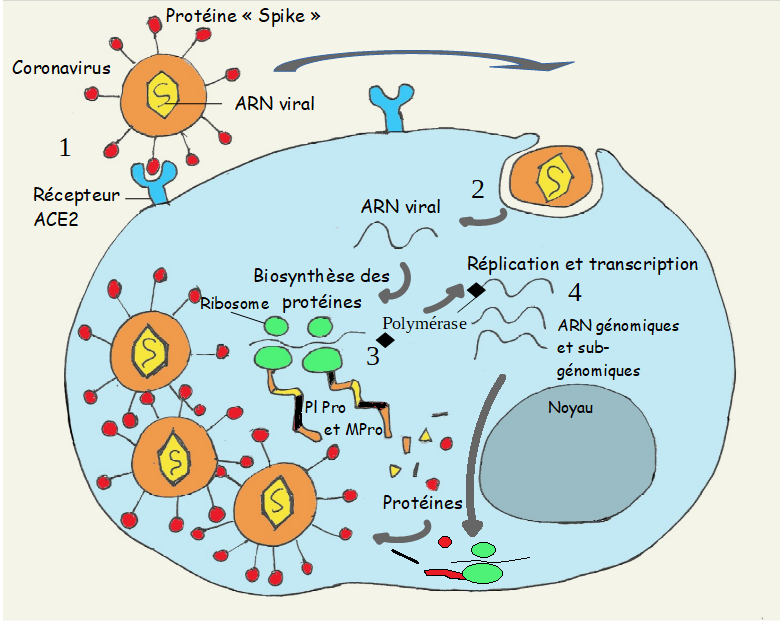
Schéma du cycle d’un coronavirus réalisé par Laure Willhelm, modifié d’après Jared S. Morse+ , Tyler Lalonde + , Shiqing Xu, and Wenshe Ray Liu . Learning from the Past: Possible Urgent Prevention and Treatment Options for Severe Acute Respiratory Infections Caused by 2019-nCoV. ChemBioChem. 2020.
Document 2 : La cristallographie permet l’identification d’antiviraux potentiels
Document 3 : Étude de l’action inhibitrice in-vitro de quelques antiviraux sur la protéase Mpro
Action de quelques inhibiteurs, testés in-vitro, sur la protéase Mpro et sur la réplication du coronavirus :


| a : Quantification du nombre de copies d'ARN viral (par ml) dans la culture cellulaire 72 h après l'infection. Mock est un témoin négatif (sans virus), COVID-19 virus est un témoin où on a infecté les cellules par le virus mais aucun inhibiteur n’a été ajouté. On utilise une concentration de 10 μM (micromoles) d’inhibiteur. b,c : courbes montrant l’action inhibitrice de l’Ebselen et de N3 sur l’infection des cellules par le SARS-CoV-2 pour différentes concentration de l’inhibiteur. (à 100 % : pas de destruction cellulaire liée à l’infection). d, e : courbes montrant l’action inhibitrice de l'Ebselen et du Disulfiram sur l’activité enzymatique de Mpro pour 0.2 μM d’enzyme, 20 μM de substrat et l’inhibiteur à différentes concentrations. |
D’après Zhenming Jin et al. Structure of Mpro from COVID-19 virus and discovery of its inhibitor. Nature. 2020.
1) Déterminez quelles sont les phases clefs du cycle viral et les protéines du SARS-CoV-2 contre lesquelles peuvent agir les antiviraux.
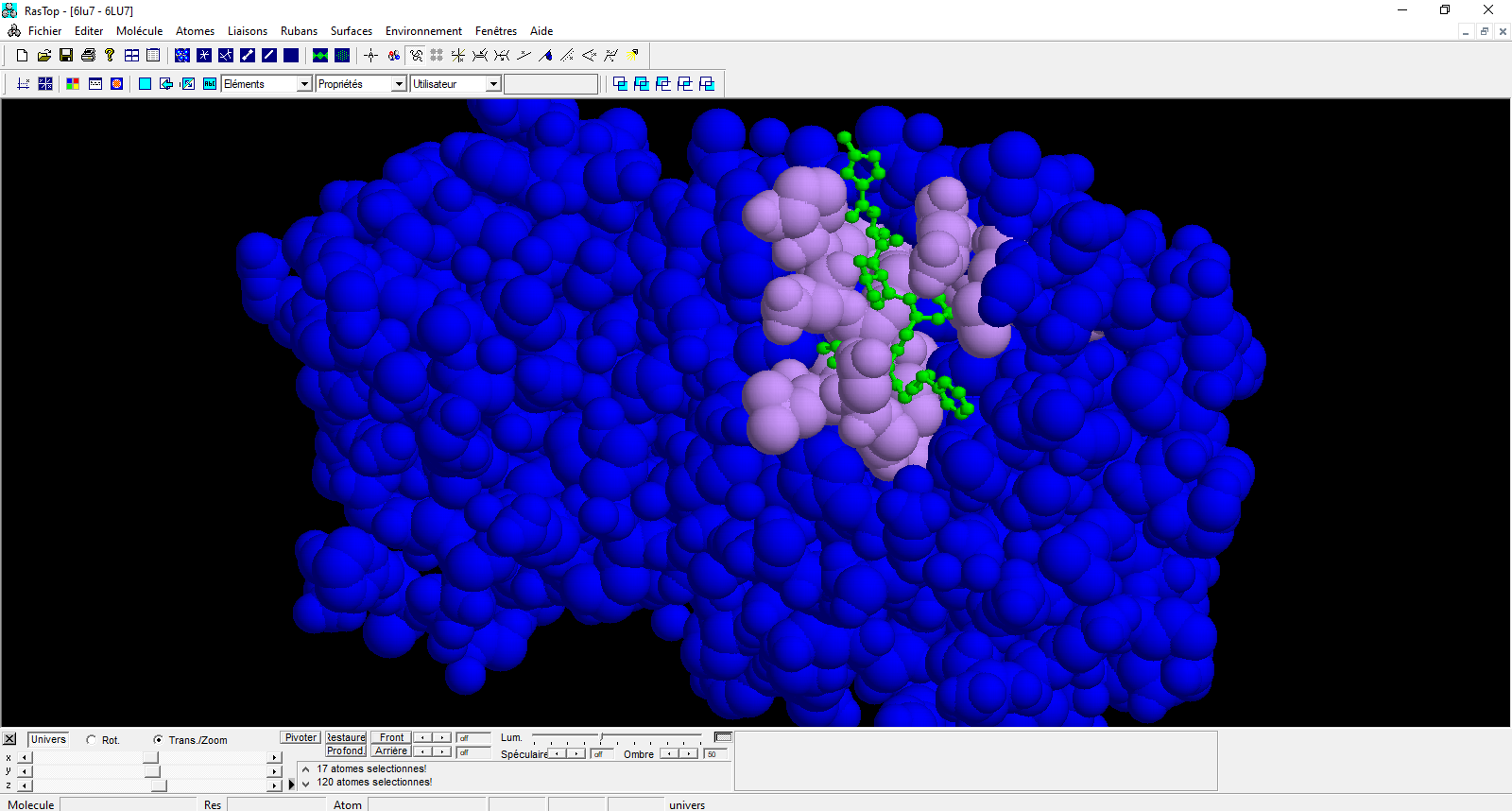
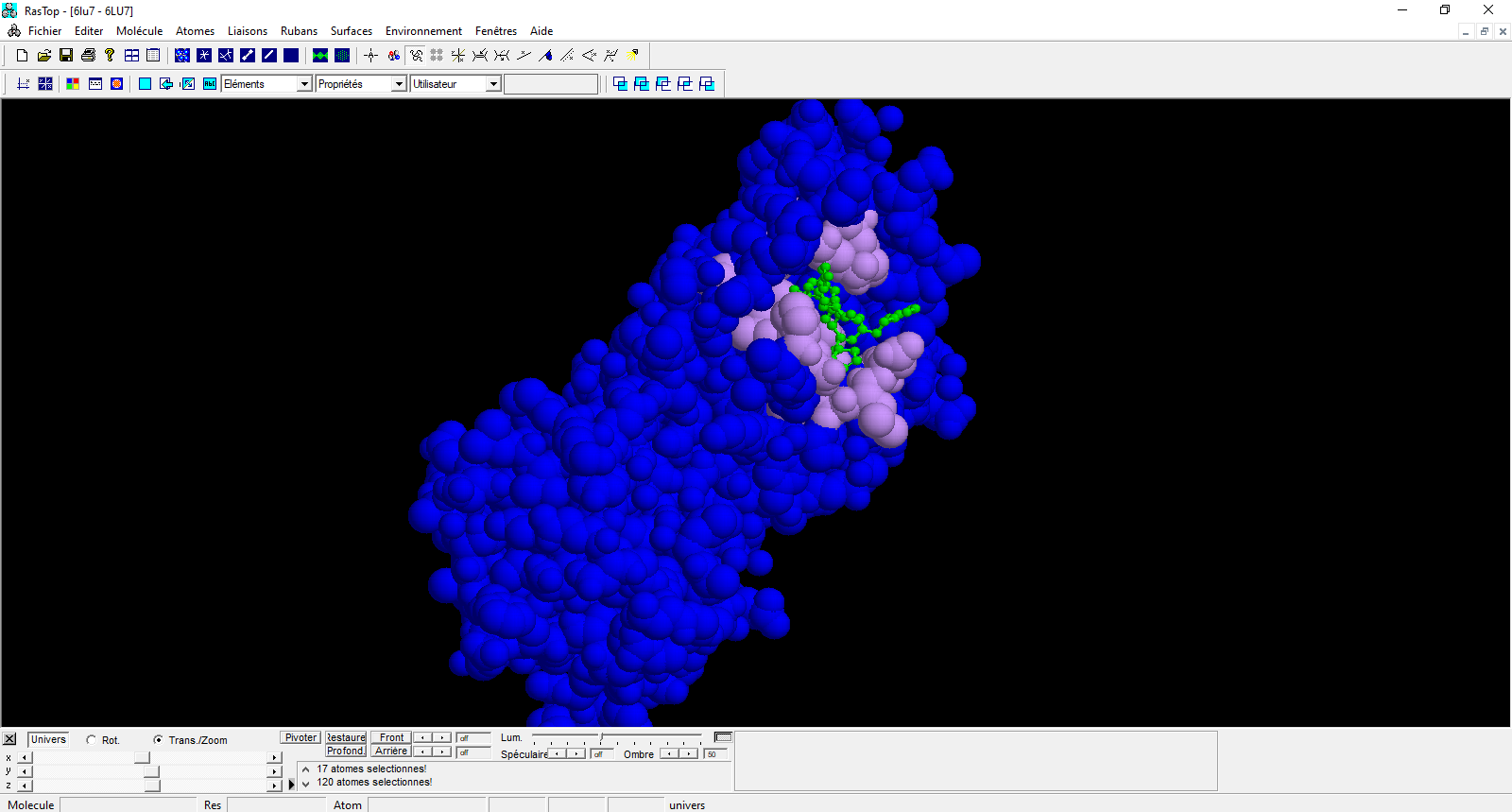
2) Ouvrez le fichier « N3-Mpro » dans Rastop afin de visualiser le modèle moléculaire en 3D de l’enzyme Mpro lié à un inhibiteur N3. Affichez l’enzyme en sphères et l’inhibiteur N3 en boules et bâtonnets, puis colorez l’inhibiteur N3 en vert et l’enzyme Mpro en bleu. Mettez en évidence en les colorant en violet les acides-aminés du site actif de l’enzyme : Ser16, Met49, Tyr54, Phe140, Leu141, Asn142, Gly143, Cys145, His163, His164, Met165, Glu166, Leu167, Pro168, Phe185, Asp187, Gln189.
Après avoir présenté de façon numérique votre modèle moléculaire légendé et titré, mettez en relation vos observations avec vos connaissances sur les enzymes pour proposer une explication à l’action inhibitrice de l’antiviral N3 sur l’activité de l’enzyme Mpro du SARS-CoV-2.
Pour télécharger le fichier N3-Mpro : N3-Mpro.pdb
3) A partir du document 3, proposez des antiviraux qui apparaissent prometteurs pour lutter contre le SARS-CoV-2 d’après l’étude de leur action sur les cellules infectée in-vitro (en laboratoire).
4) Exploitez l’ensemble des documents pour expliquer d’une part comment sont identifiés des antiviraux potentiellement prometteurs contre le SARS-CoV-2 et d’autre part comment peuvent agir ces antiviraux contre l’infection virale.
Exemples de résultats obtenus sur Rastop :