Le coronavirus SARS-CoV-2, à l'origine de la COVID-19
Une veille documentaire spécifique sur l'épidémie de COVID-19, causée par le coronavirus SARS-CoV-2, est réalisée par l'équipe ACCES. Elle liste des sites rendant compte de la situation nationale et mondiale sur le coronavirus jour après jour, des dossiers scientifiques émanant de grands organismes de recherche ainsi que des articles de vulgarisation. Cette veille est mise à jour très régulièrement.
L'objet de la présente page est de donner quelques éléments clés au sujet du virus SARS-CoV-2 lui-même.
Symptômes, traitements et transmission interhumaine
L'infection par ce nouveau coronavirus se traduit par des symptômes proches de ceux d’une grippe : fièvre, signes respiratoires comme la toux, douleurs musculaires et fatigue, avec une période d’incubation* pouvant aller de 2 à 14 jours. Dans les cas plus graves, l’infection peut entraîner une pneumonie, un syndrome respiratoire aigu sévère (SRAS), une insuffisance rénale, voire la mort. A ce jour, les cas les plus graves semblent concerner les individus les plus vulnérables (personnes âgées, personnes souffrant de maladies associées). La maladie causée par le SRAS-CoV-2 est appelée COVID-19.
Il n’existe pas de traitement spécifique contre la maladie. Certains traitements spécifiques sont à l’étude et seront testés dans le cadre d’essais cliniques. Cependant, de nombreux symptômes peuvent être traités.
Il n’existe pas de vaccin contre ce nouveau coronavirus.
Le coronavirus peut se transmettre d’une personne à une autre, généralement par contact étroit avec une personne infectée par le biais de gouttelettes respiratoires produites lors des toux et éternuements ou par des gouttelettes de salive ou des sécrétions nasales. Le coronavirus survit jusqu’à 3 heures dans le milieu extérieur, sur des surfaces inertes sèches. En milieu aqueux, ce virus peut survivre plusieurs jours.
Il est important que chacun observe de bonnes règles d’hygiène respiratoire : se couvrir la bouche et le nez avec le pli du coude ou avec un mouchoir en cas de toux ou d’éternuement et jeter le mouchoir immédiatement après dans une poubelle fermée. Il est également très important de se laver les mains régulièrement avec une solution hydroalcoolique ou à l’eau et au savon.
Ce virus est bien plus contagieux que celui du SRAS ou du MERS (syndrome respiratoire du Moyen-Orient), mais semble moins virulent, avec un taux de mortalité moindre :
-
SARS-CoV2 : 2 %
-
SARS-CoV : 10 %
-
MERS-CoV : 37 %
Les coronavirus
Les coronavirus sont des virus à ARN très répandus chez les mammifères et les oiseaux. Leur nom provient de la conformation des spicules* qui recouvrent les particules virales, leur donnant un aspect de couronne.

A. Des particules libres du virus SARS-CoV2 photographiées en microscopie électronique en coloration négative.
B. Les particules virales à la surface des cellules de l’épithélium respiratoire des patients infectés sont désignés par des têtes de flèches. Source : Zhu et al. NEJM, 2020 (DOI:10.1056/NEJMoa2001017)
La première identification d’un coronavirus remonte à 1931 aux États-Unis, avec le coronavirus responsable de la bronchite infectieuse aviaire. Depuis, de très nombreux autres coronavirus ont été identifiés et sont aujourd’hui classé en 4 groupes :
-
Alphacoronavirus
-
Betacoronavirus
-
Gammacoronavirus
-
Deltacoronavirus.
Les betacoronavirus sont pour le moment les seuls coronavirus décrits pour être à l’origine de zoonoses*. Ainsi, Le SARS-CoV-2 est un betacoronavirus comme ceux responsables du SRAS et du MERS (syndrome respiratoire du Moyen-Orient).
La dernière épidémie sévère de SRAS remonte à 2003, touchant la Chine et le Canada. Dans ce cas, les chauves-souris étaiten les animaux réservoirs. Concernant le MERS, l’épidémie date de 2012, et a touché principalement le Moyen-Orient. L’animal réservoir était le dromadaire.
Du point de vue évolutif, les coronavirus de type alpha et beta semblent provenir des chauves-souris. Les types gamma et delta proviendraient eux des oiseaux :
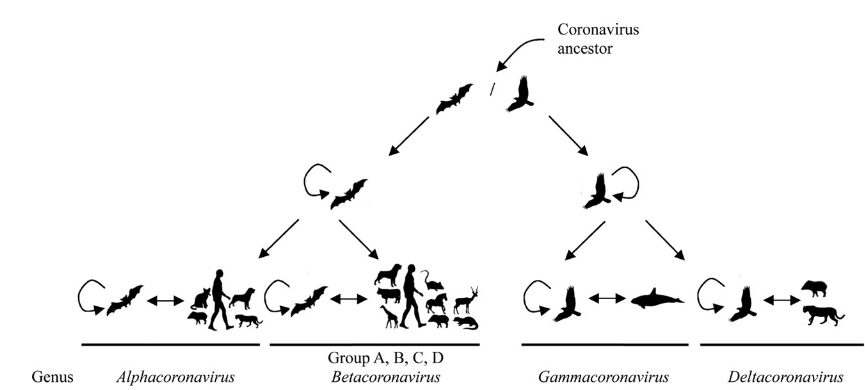
Évolution des coronavirus et réservoirs animaux. Source : Woo et al. Journal of Virology, 2012 (DOI:10.1128/JVI.06540-11)
L’origine animale du SARS-CoV-2 établie à partir des analyses génomiques
Le virus SARS-CoV-2, responsable chez l'Homme de la maladie COVID-19, est issu de l'évolution de virus initialement présents chez d'autres espèces animales. Ce virus a passé la barrière d'espèce et circule aujourd'hui de façon pandémique par transmission inter-humaine. Des études génomiques sont menées actuellement pour retracer le parcours évolutif du virus SARS-CoV-2 et identifier l’espèce animale de laquelle il a émergé (hôte source naturel, aussi appelé hôte réservoir), ainsi que de potentiels hôtes intermédiaires qui auraient facilité le passage du virus à l'Homme.
Remarque : Un hôte source naturel ne présente généralement pas de signes cliniques significatifs et visibles associés à l'infection, le virus et l'hôte ayant une longue histoire de co-évolution. Les hôtes intermédiaires peuvent eux en présenter.
De nombreuses espèces sauvages sont des hôtes de coronavirus proches du SARS-CoV-2. Les recherches génomiques s'appuient sur la comparaison des séquences du génome du SARS-CoV-2 et des virus apparentés (appelés SARSr-CoV pour SARS-related coronaviruses) identifiés chez ces espèces hôtes. Plus la séquence du SARS-CoV-2 est similaire à celle d'un SARSr-CoV identifié chez un hôte donné, plus la probabilité que le SARS-CoV-2 ait émergé à partir de cet hôte est élevée.
Il apparaît que le coronavirus humain SARS-CoV-2 présente des similitudes importantes avec deux coronavirus animaux :
- Le virus Bat SARSr-CoV RaTG13 décrit chez une espèce de chauve-souris (Rhinolophus affinis)
- Le Pangolin-CoV identifié chez une espèce de pangolin (Manis javanica).
Ce type d'analyses est compliqué par le fait que les virus peuvent évoluer non seulement par mutation d'un virus ancestral, mais aussi par recombinaison entre virus ancestraux distincts. Ainsi, les génomes viraux peuvent être des mosaïques, avec des régions issues d'un ancêtre donné et d'autres régions issues d'un autre ancêtre. Selon les régions génomiques considérées, l'histoire évolutive retracée par les études phylogénétiques sera donc différente.
Les analyses réalisées sur différentes régions du génome du SARS-CoV-2 suggèrent qu’il y a eu des recombinaisons entre les différents coronavirus étudiés, et que le génome du SARS-CoV-2 est une mosaïque de régions héritées d'un ancêtre de type Pangolin-CoV, et d'autres régions héritées d'un ancêtre de type Bat SARSr-CoV RaTG13.
La chauve-souris comme hôte source et le pangolin comme hôte intermédiaire est donc une piste très solide issue de ces études génomiques. Elle est appuyée par le fait que :
- les chauves-souris et les pangolins partagent des niches écologiques qui se chevauchent,
- et que les pangolins infectés présentent des signes cliniques similaires à ceux du COVID-19, ce qui est cohérent avec un rôle du pangolin comme hôte intermédiaire plutôt que comme hôte source.
Comment expliquer la "réussite évolutive" du SARS-CoV-2 ?
Le potentiel d'un virus à passer la barrière d'espèce et à se disséminer ensuite au sein de la population humaine, est lié entres autres à son équipement protéique lui permettant (ou non) d'infecter des cellules humaines. Les protéines de surface du virus sont importantes pour l'entrée du virus dans ses cellules cibles, car elles permettent la liaison du virus à des protéines cellulaires qui se comportent comme des récepteurs pour le virus. Le récepteur cellulaire du SARS-CoV-2 est la protéine membranaire ACE2, une enzyme de conversion de l’angiotensine 2. Le virus s’y associe via les protéines Spike (codées par le gène S), présentes à la surface des particules virales, et plus particulièrement via le domaine dit "RBD" de liaison aux récepteurs (receptor-binding domain).
Au sein de la mosaïque génomique du virus SARS-CoV-2, la séquence du domaine RBD de la protéine Spike ne présente qu’une seule différence d’acide aminé avec celle du Pangolin-CoV. Cela suggère que la séquence du RBD a été héritée d'un ancêtre de type Pangolin-CoV. Le RBD des virus de type Pangolin-CoV semble donc particulièrement adapté pour interagir avec le récepteur ACE2 humain, ce qui confèrerait alors le grand potentiel d'émergence au virus recombinant SARS-CoV-2.
Pour expliquer cela, l'hypothèse selon laquelle les protéines ACE2 humaine et de pangolin seraient similaires dans la région d'interaction avec le RBD de la protéine Spike des coronavirus a été formulée. Cependant, cette hypothèse ne semble pas supportée par les données disponibles. D'autres analyses sont donc nécessaires pour mieux comprendre en quoi un RBD de type Pangolin-CoV pourrait participer à la réussite évolutive du SARS-CoV-2.
Par ailleurs, la protéine Spike du SARS-CoV-2 présente une autre caractéristique absente des virus de chauve-souris et de pangolin apparentés : elle présente un site de clivage particulier entre les deux sous-unités S1 et S2 de la protéine Spike. Il est donc possible que d'autres virus d'espèces sauvages, non encore identifiés, aient participé à l'apparition du virus SARS-CoV-2.
En conclusion, les dernières recherches suggèrent que le virus SARS-CoV-2 pourrait trouver son origine dans des évènements de recombinaison entre de multiples virus d'espèces animales sauvages, faisant intervenir au moins un virus de chauve-souris (la chauve-souris serait alors l'hôte source) et un virus de pangolin (le pangolin serait alors un hôte intermédiaire).
Cependant, certaines similarités de séquences pourraient également être expliquées par des scénarios alternatifs, basés par exemple sur la convergence évolutive. Ces scénarios alternatifs ne peuvent pour le moment être exclus.
Lexique
Spicules (Spike) : glycoprotéines de surface permettant au virus de reconnaître la cellule-cible.
Période d’incubation : c’est le temps qui s’écoule entre l’infection et l’apparition des symptômes cliniques de la maladie.
Zoonose : maladie infectieuse des vertébrés transmissibles à l'être humain (ex. la rage).
Sources
https://www.sciencemag.org/tags/coronavirus
https://www.sciencemag.org/news/2020/01/mining-coronavirus-genomes-clues-outbreak-s-origins
http://www.afas.fr/epidemie-de-wuhan-virus-proche-a-96-coronavirus-present-chez-chauve-souris/
https://www.biorxiv.org/content/10.1101/2020.01.22.914952v2
https://www.nature.com/articles/s41586-020-2313-x_reference.pdf
https://www.nature.com/articles/s41586-020-2169-0_reference.pdf
http://www.zoores.ac.cn/article/doi/10.24272/j.issn.2095-8137.2020.031
https://jvi.asm.org/content/early/2020/05/14/JVI.00831-20.long
