Glossaire
Histoire
Téléchargements
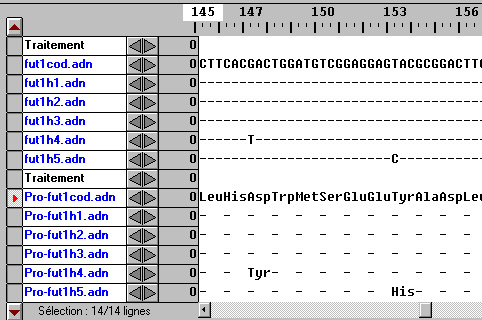
Comparaison des allèles de Fut 1 et des polypeptides correspondants
(L'allèle fut1cod.adn code pour une enzyme EH fonctionnelle ; tous les autres allèles codent pour une enzyme EH non fonctionnelle)
Alignement simple

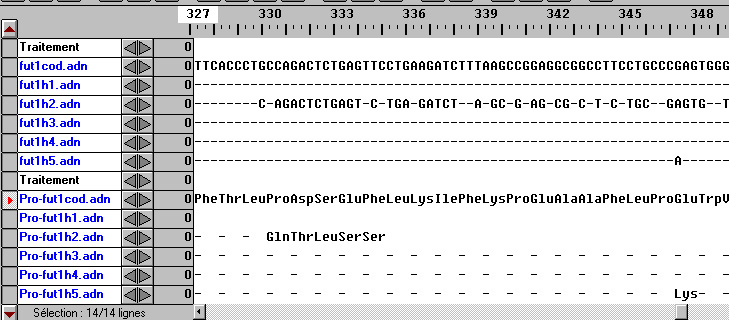


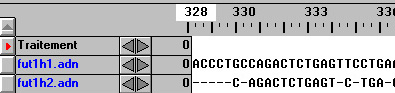
Alignement avec discontinuité
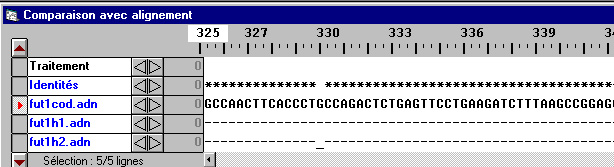
- Il y a une délétion dans l'allèle fut1h2 (disparition du nucléotide G), ce qui entraîne le décalage du cadre de lecture du gène. Il y a alors apparition précoce d'un codon stop (336e codon) :
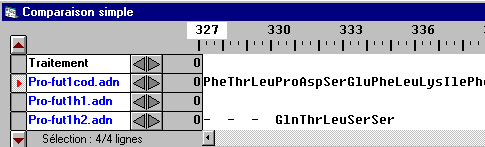
(comparaison simple)
Les allèles fut1h1 et fut1h2 codent pour des polypeptides beaucoup plus courts que la normale : les mutations (délétion pour fut1h2, substitution pour fut1h1) font apparaître des codons stop. L'enzyme ne peut donc pas prendre sa conformation spatiale normale et elle est donc non fonctionnelle. Les allèles fut1h3, fut1h4 et fut1h5 codent pour des polypeptides qui diffèrent de l'enzyme fonctionnelle par quelques acides aminés seulement. Ces polypeptides correspondant à des enzymes non fonctionnelles, on peut supposer que les acides aminés modifiés se situent au niveau du site actif de l'enzyme, ou qu'ils participent de façon importante à la structure spatiale de l'enzyme.