 |
SEQAID II - Minidoc |
 |
SEQAID II - Minidoc |
Exemple de thème
Détermination du génotype
des individus à risque
grâce à la méthode
des enzymes de restriction
Niveau : Terminale S, Enseignement de spécialité
Partie du programme : Application et implications des connaissances modernes en génétique humaine.
L'exemple traité est celui d'une forme d'albinisme oculomoteur.
Les réponses sont données en italique et en vert
Arbre généalogique de la famille :
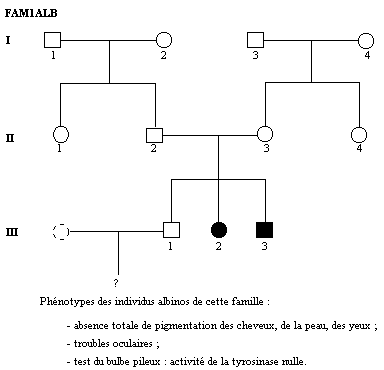 |
Le risque pour le couple III0 x III1 d'avoir un enfant atteint d'albinisme, sachant que la fréquence des hétérozygotes dans la population est de 1%, est de :
2/3 x 1/100 x 1/4 = 1/600.Le diagnostic génétique n'est pas évident à établir par cette méthode classique car il ne donne qu'une probabilité de risque d'apparition d'une maladie sans pouvoir donner une réponse avec certitude.
Caractéristiques génétiques de l'albinisme oculomoteur de type 1 :
On repère une seule différence au niveau de la base 575.
Ainsi la base adénine A dans l'allèle TYRCOD1 devient cytosine C dans l'allèle TYRCOD2
Il s'agit d'identifier l'enzyme spécifique, pour cela on dispose d'une banque de données sur les enzymes de restriction dans le fichier ENZTYR.
| Noms des enzymes de restriction | Motifs reconnus |
| Msp I | CCGG |
| Mnl I | CCTC |
| Hae III | GGCC |
| EINV2 | CAATC |
| Xho II | GGATCT |
| Xba I | TCTAGA |
| EINV4 | AGTGTG |
La première recherche proposée
informe qu'au niveau des bases 571 à 576 de l'allèle TYRCOD2
on trouve la séquence GGATCT. La seule enzyme capable de couper
dans la région 575 est Xho II.
Vérification :
Résultat de l'action de Xho II sur les 2 allèles fonctionnels :
| Allèles sélectionnés et comparés | Positions des sites reconnus | Longueurs des fragments obtenus |
| TYRCOD1 | 1135, 1418 | 172, 283, 1135 |
| TYRCOD2 | 571, 1135, 1418 | 172, 283, 564, 571 |
On peut représenter schématiquement les sites de coupure de l'enzyme de restriction testée sur chaque allèle, identifier et visualiser les différents fragments de restriction et imaginer le résultat de l'électrophorèse des différents fragments obtenus.
Sur le schéma de l'ADN ci-dessous (qui servira de modèle) on a placé la position de bases repères depuis la position n°1 jusqu'à la dernière base n°1590 :
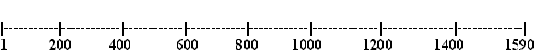 |
Sur le schéma, les sites de coupure
réalisés par l'enzyme spécifique XhoII sur
chaque allèle sont désignés par :Ê
et les fragments de restriction sont mentionnés.
 |
| Noms des enzymes de restriction | Motifs reconnus | positions des sites reconnus sur TYRCOD1 | positions des sites reconnus sur TYRCOD2 |
| Msp I | CCGG | 2 | 2 |
| Mnl I | CCTC | 11 | 11 |
| Hae III | GGCC | 7 | 7 |
| EINV2 | CAATC | 0 | 0 |
| Xho II | GGATCT | 2 | 3 |
| Xba I | TCTAGA | 0 | 0 |
| EINV4 | AGTGTG | 1 | 1 |
II - Reconnaissance de tous les allèles du gène de la tyrosinase par la construction d'un tableau de comparaison ou de référence
On peut appliquer la méthode vue dans le paragraphe précédent à tous les allèles du gène de la tyrosinase notamment les allèles non fonctionnels.
| Enzymes de restriction | Msp I | Mnl I | Hae III | EINV2 | Xho II | Xba I | EINV4 |
| Allèles à comparer | |||||||
| TYRCOD1
(référence 1) |
|||||||
| TYRCOD2
(référence 2) |
|||||||
| TYRALBA1 | |||||||
| TYRALBA2 | |||||||
| TYRALBA3 | |||||||
| TYRALBA4 | |||||||
| TYRALBA5 |
| Enzymes de restriction | Msp I | Mnl I | Hae III | EINV2 | Xho II | Xba I | EINV4 |
| Allèles à comparer | |||||||
| TYRCOD1
(référence 1) |
|
|
|
|
|
|
|
| TYRCOD2
(référence 2) |
|
|
|
|
|
|
|
| TYRALBA1 |
|
|
|
|
|
|
|
| TYRALBA2 |
|
|
|
|
|
|
|
| TYRALBA3 |
|
|
|
|
|
|
|
| TYRALBA4 |
|
|
|
|
|
|
|
| TYRALBA5 |
|
|
|
|
|
|
|
III - Détermination des génotypes des individus de l'arbre généalogique
Nombre de sites reconnus par les différentes enzymes
| Individus | Allèles du gène | Allèles du gène des individus |
|
|
|
|
|
|
|
| III1 | III1 ALL1 | TYRCOD2 | 2 | 11 | 7 | - | 3 | - | 1 |
| III1 | III1 ALL2 | TYRCOD2 | 2 | 11 | 7 | - | 3 | - | 1 |
Il est donc homozygote sain.
Comme III1 est homozygote avec 2 allèles fonctionnels, il ne transmettra pas d'allèle muté.
Dans ce cas même, si son épouse IIIo est hétérozygote le génotype double récessif ne pourra pas se constituer, ainsi le couple IIIo x III1 ne risque pas d'avoir un enfant albinos.
Conclusion :
L'analyse génotypique permet d'affiner et de préciser les prévisions qui sont habituellement faites d'après l'étude de l'arbre généalogique qui est basée sur l'examen des phénotypes.
Cela est possible grâce à l'utilisation des enzymes de restriction qui permettent de connaître indirectement la nature des allèles.