Glossaire
Histoire
Téléchargement
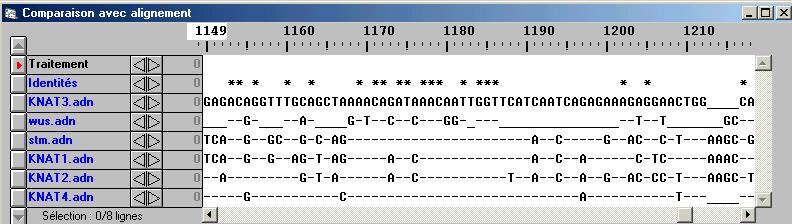
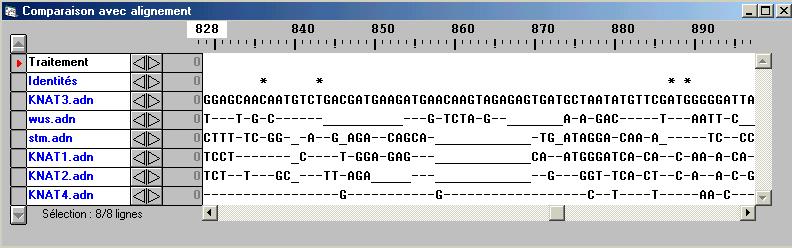
On réalise ci-dessous, à l'aide du logiciel Anagène, un alignement avec discontinuité des séquences nucléotidiques des gènes WUS, STM et de divers gènes KNUT. Il s'agit des gènes dont les mutations engendrent respectivement les phénotypes mutés Wus, Stm et Knotted. Le gène CLV dont la mutation est responsable du phénotype muté Clavata est volontairement exclu de cette comparaison, car il ne s'agit pas d'un gène à homéoboite : le gène CLV code pour une protéine kinase de type récepteur et non pour une protéine à homéodomaine.
L'image ci-dessous montre le résultat de la comparaison des séquences, observé dans une portion de séquence ne correspondant pas à l'homéoboite : la faible densité des astérisques (on rappele qu'un astéristque correspond à un nucléotide identique dans toutes les séquences alignées) souligne le peu de ressemblances entres ces portions de séquences :
L'image ci-dessous montre le résultat de la comparaison des séquences, observé dans une portion de séquence faisant certainement partie de l'homéoboite : les astérisques sont plus nombreuses et plus rapprochées, ce qui souligne le fort degré de ressemblance entre ces séquences :