Glossaire
Histoire
Téléchargements
Classer des animaux Vertébrés (6ème)
Etablir des relations de parenté :
En Tle S, à partir de données moléculaires
à partir de données moléculaires (Tle S)
Proposée par Monique Dupuis, Lycée
Jean Monnet, La-Queue-les-Yvelines,
et Jean-Claude Hervé
Démarche basée sur l'utilisation du logiciel
Phylogène.
L'établissement de relations de parenté entre les organismes peut se faire à partir de données moléculaires (protéiques ou nucléiques), en utilisant au lycée la méthode phénétique. La premier étape consiste à introduire la notion de molécules homologues.
Des séquences alignées relatives aux sont disponibles dans la rubrique Téléchargements.
- Notion de molécules homologues
- Utilisation d'une méthode phénétique pour établir les relations de parenté entre ces organismes à partir des données moléculaires fournies
Pour pouvoir dégager la notion de molécules homologues, il faut disposer de séquences alignées.
Exemple : séquences alignées des protéines codées par le gène CDC2 contrôlant l'entrée en mitose du cycle cellulaire, chez diverses espèces (seuls les débuts des séquences de ces molécules sont représentées ici).
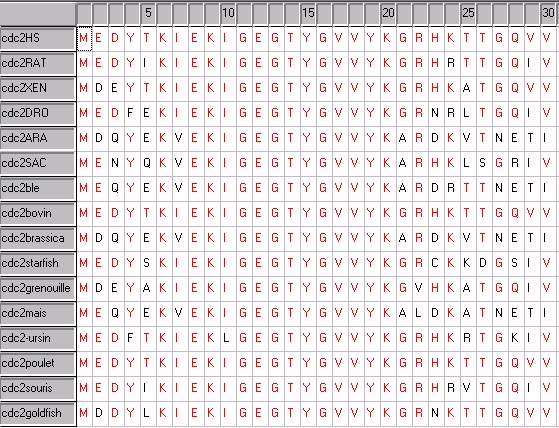
Conclusion à tirer de l'observation des séquences alignées : le constat de très nombreuses similitudes dans les séquences de ces molécules permet de dire qu'elles doivent avoir une origine commune; on parle alors de molécules homologues. On peut donc en déduire que les organismes qui possèdent ces molécules ont également une origine commune (leur ancêtre commun possédait la molécule à l'origine de toutes celles qui sont observées ici).
II)
Utilisation d'une méthode phénétique pour établir
les relations de parenté entre ces organismes à partir des
données moléculaires fournies
Les méthodes phénétiques sont basées sur
les imilitudes globales, sans tenir compte des états dérivés
ou primitifs des caractères.
Il faut donc établir une matrice des différences (ou
des similitudes) entre les molécules étudiées.
L'observation du tableau de comparaison des séquences révèle que le nombre de différences entre une séquence et celle qui est prise comme référence varie selon les organismes. Le logiciel Phylogène permet d'obtenir la matrice des distances entre les séquences des divers organismes prises deux à deux.
Une approche progressive pourrait être la suivante :
- Acquisition du principe de construction d'un arbre
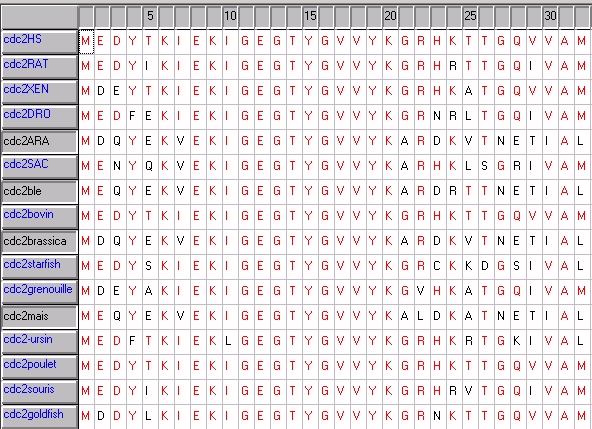
Dans un premier temps, il est souhaitable de se limiter à un petit nombre d'espèces (par exemple aux quatre espèces végétales, mais toutes les stratégies de choix d'espèces sont possibles). Dans le tableau d'alignement des séquences, en cliquant sur le nom d'une espèce, celui-ci se colore en bleu, signifiant que la séquences est exclue du calcul (en recliquant sur le nom d'une séquence exclue, on l'intégre à nouveau dans le calcul).
.
Ensuite, on peut demander l'affichage de la matrice des distances pour les espèces retenues (Exprimées en poucentage des différences).
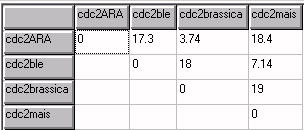
La réflexion sur les données de cette matrice conduit à l'idée que, plus il y a de différences entre deux molécules et plus la molécule ancestrale commune est éloignée dans le temps, donc plus les êtres vivants qui possèdent ces molécules ont un ancêtre commun éloigné dans le temps (et inversement).
Les élèves sont conduits à traduire ces relations de parenté par un arbre, puis de confronter ce dernier avec celui fourni par le logiciel (Cliquer sur Arbre). Les méthodes de construction d'arbres, utilisées par les spécialistes (UPGMA, NJ) n'étant pas au programme, il ne s'agit ici que d'une représentation qualitative des relations de parenté.
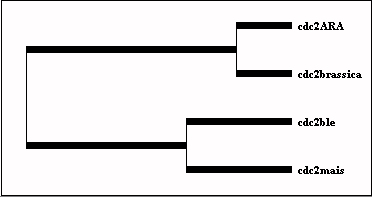
On peu rajouter une ou deux espèces, afficher une nouvelle matrice, inviter à construire le nouvel arbre, et à nouveau le confronté à celui calculé par le logiciel.
Cet apprentissage de la méthode de construction d'un arbre, recommandé par les instructions, prépare à la lecture d'arbres établis à partir de données moléculaires.
- Lecture et interprétation d'un arbre avec l'ensemble des espèces
On peut maintenant considérer l'ensemble des espèces, demander la matrice et l'arbre correspondants.
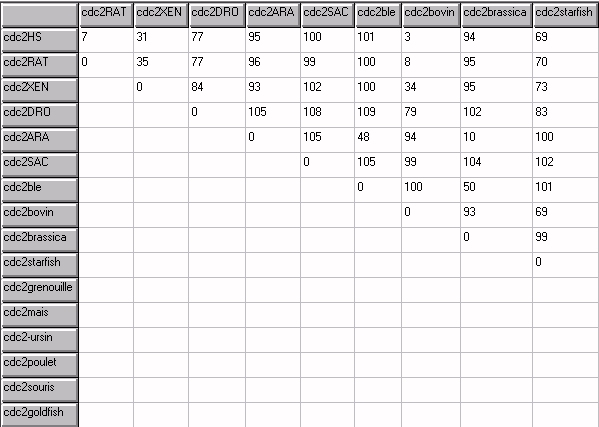
Matrice partielle des séquences de CDC2 obtenue avec Phylogène
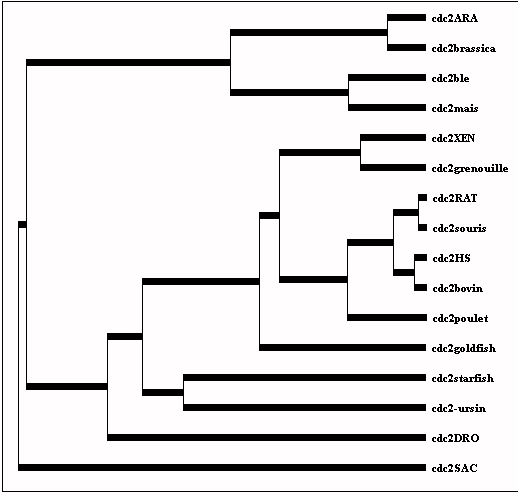
Arbre correspondant à la matrice précédente
tracé par Phylogène.
Il convient à ce stade de voir si les informations tirées de la lecture de cet arbre concordent avec les connaissances sur les relations de parenté établies à partir des données anatomiques et morphologiques.
C'est une première validation de l'usage des données moléculaires pour établir des relations de parenté entre espèces.
- Cohérence entre les résultats fournis par les séquences de différetes protéines
La confrontation de cette matrice et de cet arbre avec ceux obtenus avec Phylogène à partir des chaînes alpha et bêta de globine, fournit une autre occasion pour discuter de la validité des données moléculaires.
La pertinence des données moléculaires étant globalement
établies, cela justifie l'exploitation de ce type de données
pour déterminer les relations de parenté au sein des Primates
hominoïdes, alors que les données anatomiques et morphologiques
sont inexploitables au lycée.