Glossaire
Histoire
Téléchargements
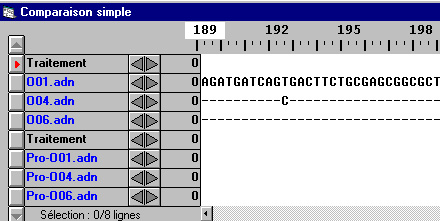
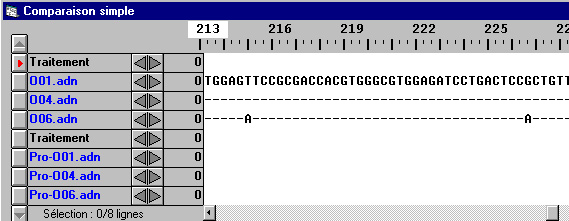
- Comparaison par alignement simple des allèles et des polypeptides A
- Comparaison par alignement simple des allèles et des polypeptides B
- Comparaison par alignement simple des allèles et des polypeptides O
Comparaison par alignement simple des allèles A et des polypeptides correspondants
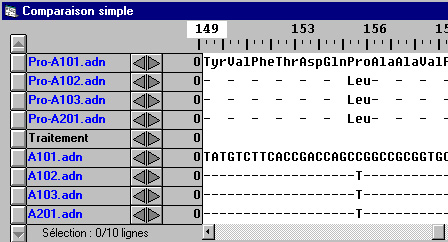
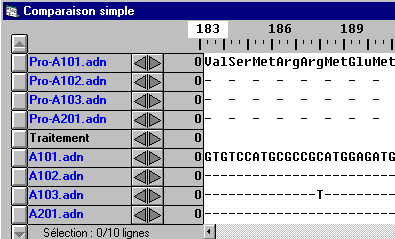

(il y a 1128 nucléotides pour A201)
Comparaison par alignement simple des allèles B et des polypeptides correspondants
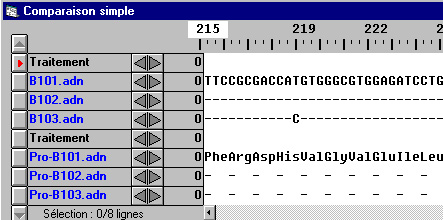
(tous les allèles B présentés ici ont 1065 nucléotides)
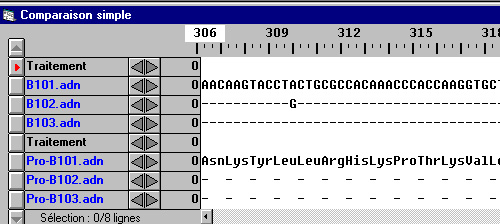
Les allèles B101, B102 et B103 codent tous pour des enzymes EB fonctionnelles, ayant le même rôle. Les acides aminés modifiés dans la séquence protéique ne doivent donc pas intervenir de façon importante dans la structure spatiale de la molécule, et ne doivent pas être situés au niveau du site actif. Il y aura donc toujours synthèse possible du marqueur B à partir de H.
Comparaison par alignement simple des allèles O et des polypeptides correspondants
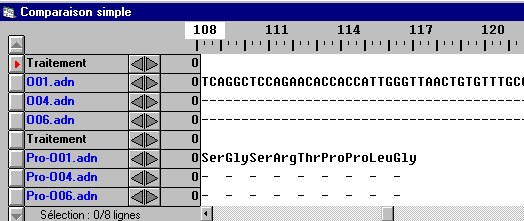
(aucune différence de séquence de nucléotides pour les 118 premiers codons)