|
|
|||||||||
|
Démarche proposée par Monique Dupuis, Lycée J. Monnet, La Queue-Les-Yvelines En classe de Tle S, on repart de ce qui a été établi en classe de 4e, notamment en ce qui concerne la classification des Vertébrés. Les ordres d’apparitions (et leur époque) des différentes classes de Vertébrés au cours des temps géologiques sont fournis à l’élève. Démarches correspondant au programme de Tle S actuellement en vigueur
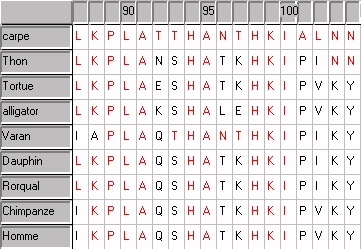
1. Etablissement de relations de parenté au sein des Vertébrés à partir de données anatomiques, morphologiques et embryologiques La démarche pédagogique applicable dans les anciens programmes de Tle S (programmes encore en vigueur cette année 2001/2002) est semblable à celle applicable dans les nouveaux programmes de 4e (relations de parenté 4e). 2. Etablissement de relations de parenté au sein des Vertébrés à partir de données moléculaires(Il ne s'agit pas ici d'entrer dans le détail des méthodes phénétiques de traitement des données moléculaires, mais juste d'en faire comprendre le principe aux élèves). a) La notion de molécules homologues et sa signification évolutive Plusieurs
organismes peuvent posséder une "même" molécule protéique
ou nucléique (exemple : tous les Vertébrés posssèdent
un gène de la myoglobine codant pour une molécule de myoglobine
ayant pour rôle la fixation de dioxygène). La comparaison
des séquences alignées de ces molécules peut fournir
des informations relatives aux parentés entre ces organismes.
Conclusion
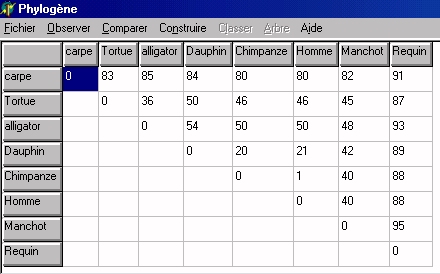
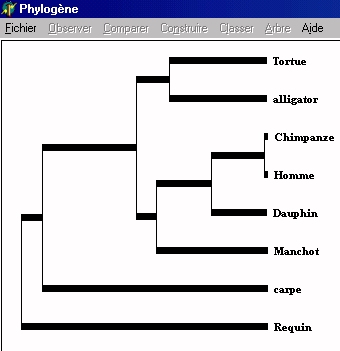
La méthode cladistique (utilisée pour traiter les données anatomiques et morphologiques) est applicable aux données moléculaires : chaque site (c'est à dire chaque position, chaque acide aminé ou chaque nucléotide) est considéré comme un caractère pouvant avoir plusieurs états (les différents acides aminés ou nucléotides que l'on peut trouver à cette position). Le problème est alors de savoir quel est l'état dérivé de chacun de ces caractères. Plusieurs hypothèses seront faites, et c'est le principe de parcimonie qui sera appliqué et qui permettra de bâtir l'arbre phylogénétique le plus probable. Cette méthode, qui nécessite des calculs nombreux par le logiciel et une discussion sur les différents arbres obtenus ne sera pas appliquée avec des élèves de Tle S. On se contentera d'aborder le traitement des données moléculaires par une méthode phénétique, basée sur le nombre global de différences, telle qu'elle est présentée ci-dessous. Deux démarches pédagogiques différentes sont proposées : Proposition 1 La
comparaison de molécules homologues appartenant à des organismes
différents permet de préciser leurs relations de parenté
si l'on se base sur le principe suivant : si l'on considère que
les mutations apparaissent (et se fixent) relativement régulièrement
au cours du temps (principe de l'horloge moléculaire), on peut alors
dire que moins le nombre de différences entre deux molécules
homologues est important et plus la molécule ancestrale dont elles
dérivent est récente, et donc plus les organismes qui possèdent
ces molécules ont un ancêtre récent. La matrice des
différences servira de base au raisonnement.
3. Etablissement de relations de parenté au sein des Vertébrés à partir de données anatomiques, morphologiques, embryologiques et moléculaires Des démarches correspondant au nouveau programme de Tle S (qui entrera en vigueur en Septembre 2002) seront bientôt mises en ligne ... En cours de réalisation... |
||||||||