Établir des parentés à partir de données moléculaires
![]() Phylogène permet de tracer un arbre à partir d'un fichier contenant des séquences nucléiques ou protéiques .
Phylogène permet de tracer un arbre à partir d'un fichier contenant des séquences nucléiques ou protéiques .
Plusieurs fichiers de séquences alignées sont fournis avec le logiciel. Quelques unes sont adaptées à l'étude de ces relations de parentés sur l'ensemble du vivant. D'autres sont appropriées pour mettre en évidence la notion de famille multigénique ainsi que l'histoire des allèles d'un gène au cours de l'évolution. Ces fichiers au format .aln ont été alignés préalablement avec le logiciel ClustalW.
Après avoir choisi la collection, on accède à ces fichiers par l'icone ![]() ou par le menu Étude moléculaire (l'accès antérieur par
ou par le menu Étude moléculaire (l'accès antérieur par  Fichier/Ouvrir a été conservé). Les fichiers alignés .alp, spécifiques de Phylogène, comportent des mots-clés qui permettent de sélectionner rapidement des groupes de séquences.
Fichier/Ouvrir a été conservé). Les fichiers alignés .alp, spécifiques de Phylogène, comportent des mots-clés qui permettent de sélectionner rapidement des groupes de séquences.
Cas des fichiers de séquences alignées
- Choisir les séquences à utiliser :
- une à une en cliquant sur leur nom :
- collectivement à l'aide du menu Sélection/ sélectionner... (les choix sont enrichis si le fichier chargé comporte des mots-clés (fichier .alp). Les mots-clés peuvent être ajoutés ou édités uniquement en mode professeur (un menu Mots-Clés est alors présent). Ils peuvent servir aussi à colorer les noms des séquences dans les arbres.
- une à une en cliquant sur leur nom :
- Choisir les options pour les calculs de la matrice et de l'arbre (menu ou bouton Options)
- Faire afficher la matrice des distances (menu Afficher/Matrice ou bouton Matrice) :
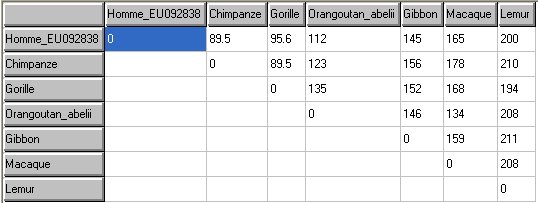
Matrice des distances de quelques Primates (nombre de différences)
- Faire afficher l'arbre correspondant (menu Afficher/Arbre ou bouton Arbre) :
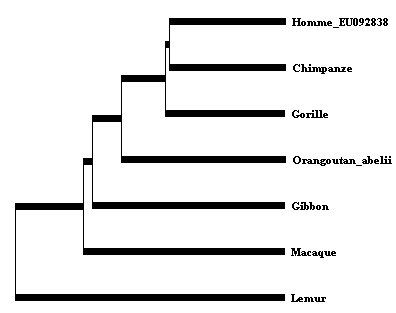
Arbre des relations de parenté de quelques Primates (UPGMA)
Cas des fichiers de séquence non alignés
C'est en particulier le cas des fichiers .edi d'Anagène qui peuvent maintenant être utilisés par Phylogène.
La première chose à faire est de demander l'alignement (bouton aligner). Contrairement à Anagène, toutes les séquences affichées (sélectionnées ou non) sont alignées. Si on veut laisser de côté certaines séquences avant l'alignement, il faut les supprimer avant de faire l'alignement (menu Édition/supprimer les séquences non séléctionnées).
Le travail est ensuite le même que celui qui a été décrit pour les séquences alignées.
Mise en couleur
- Mise en couleur des séquences
Le menu permet de colorer chaque résidu avec une couleur standard (les couleurs utilisées par ClustalX).
permet de colorer chaque résidu avec une couleur standard (les couleurs utilisées par ClustalX).
exemple :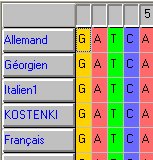
- Mise en couleur des noms des séquences en fonction de mots-clés
Lorsque le fichier contient des mots-clés (fichiers .alp), ceux-ci peuvent servir à colorer l'arbre. On repère ainsi mieux les séquences d'un groupe. La figure ci-dessous montre un arbre établi à partir de séquences de la boucle D1 de l'ADN mitochondrial de deux Hommes de Néanderthal et de quatre Hommes modernes actuels et fossiles.
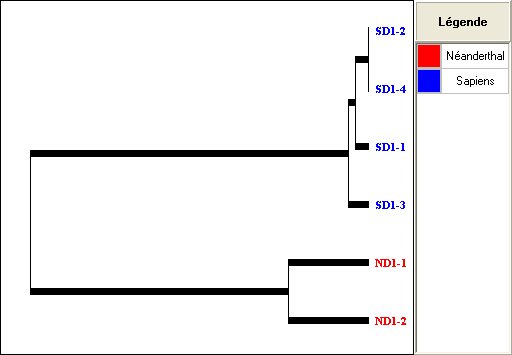
Arbre coloré avec sa légende (copié avec le menu Édition/Copier/l'arbre)
Choix concernant la matrice
Cliquer sur le menu ou le bouton Options puis choisir l'onglet Distances
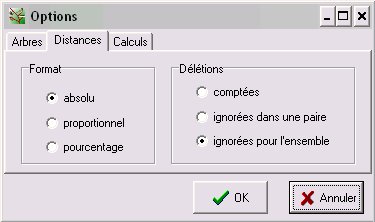
absolu : nombre de différences
proportionnel : de 0 à 1
pourcentage : de 0 à 100
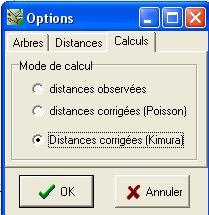
L'onglet Calculs permet de demander des corrections :
|
|
distances observées : pas de correction distance corrigée : une correction est appliquée pour tenir compte de la probabilité qu'il y ait eu 2 changements successifs à certaines positions. |
Choix concernant le calcul de l'arbre
Phylogène permet de choisir entre 3 méthodes pour construire l'arbre :
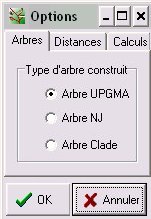 |
Arbre UPGMA : arbre le plus simple à calculer. Les calculs peuvent être vérifiés à la main pour une petite matrice. Pour être exact, il suppose que l'hypothèse de l'horloge moléculaire est vérifiée (le taux de mutations est constant dans le temps et identique pour toutes les branches), ce qui n'est pas toujours vrai. Arbre NJ (Neighbor Joining) : c'est un arbre qui n'oblige pas à faire d'hypothèse d'horloge moléculaire. La racine représentée est arbitraire et modifiable. Il faut donc utiliser un extragroupe pour enraciner l'arbre de manière raisonnée (voir ci-dessous l'exemple de la construction d'un arbre des Amniotes). Arbre clade : il faut le construire "à la main" par la méthode cladistique. |
| Permuter 2 branches au niveau d'un noeud (fonctionne avec tous les types d'arbre) | |
| Afficher les distances sur les branches (arbre UPGMA et arbre NJ) | |
| Positionner la racine de l'arbre (arbre NJ et arbre clade). |
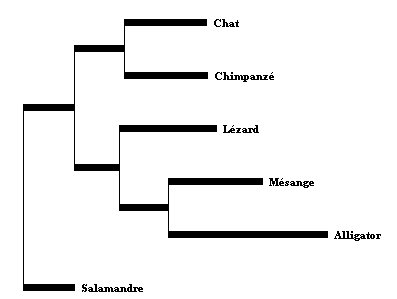
Construction d'un arbre des Amniotes
- Choisir la collection Vertébrés lycée
- Charger le "tableau de molécules" :12S-Vertebres-Etendu.aln
- Choisir les taxons Chimpanzé, Chat, Lézard, Mésange, Salamandre (extragroupe) et Alligator.
- Comme option, choisir distance en pourcentage (et éventuellement des calculs avec distances corrigées (Kimura)).
- Faire afficher la matrice des distances.
- Faire afficher l'arbre UPGMA
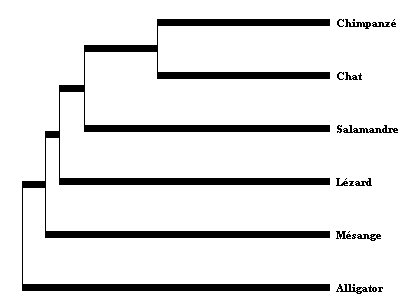 Le résultat est étonnant : la vitesse d'évolution a dû être beaucoup plus rapide dans certaines branches (Alligator en particulier)
Le résultat est étonnant : la vitesse d'évolution a dû être beaucoup plus rapide dans certaines branches (Alligator en particulier) - Choisir un Arbre NJ et le faire afficher.
Tel qu'il est enraciné, le résultat ne semble pas plus probant :
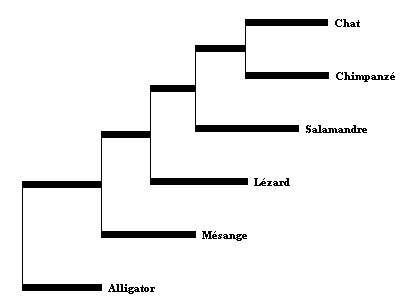
L'extragroupe n'est pas à la racine, il faut donc enraciner cet arbre (cliquer sur l'icône puis cliquer sur la Salamandre (dans l'arbre).
puis cliquer sur la Salamandre (dans l'arbre). - L'arbre obtenu correspond mieux à l'arbre cladistique que l'on peut construire à partir des états dérivés des caractères morphologiques :