Activité pédagogique sur l’apparition et la propagation de nouveaux variants du SARS-CoV-2
L’apparition et la propagation de nouveaux variants du SARS-CoV-2
Le gouvernement britannique a annoncé, à six jours de Noël, que des chercheurs avaient identifié un nouveau variant du coronavirus SARS-CoV-2. Le génome de ce variant comporte un nombre de mutations plus important que la plupart des autres variants. Le nombre des cas d’infections par ce nouveau variant, de même que le nombre de régions concernées, augmente rapidement. Ce nouveau variant est nommé VUI 202012/01 et appartient à un lignage, autrement dit à un sous-groupe de virus apparentés génétiquement, baptisé B.1.1.7. Par ailleurs, sur la base de modélisations, le gouvernement britannique a déclaré que le nouveau variant se transmettait d'individu à individu avec une efficacité jusqu'à 40 à 70% plus élevée que le virus d'origine.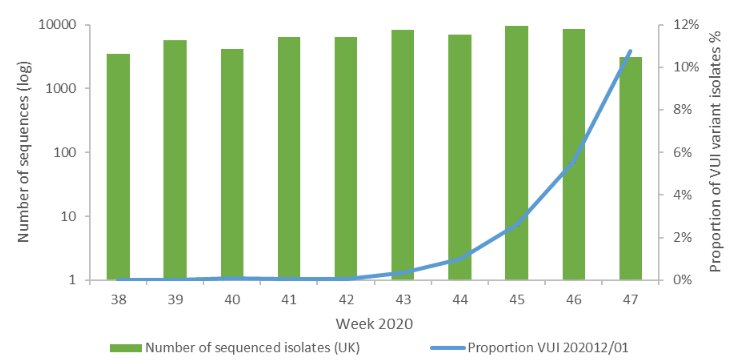
Proportion des variants VUI-202012/01 parmi tous les isolats récemment séquencés au Royaume-Uni entre la 38ème et 47ème semaine de 2020. Base de données GISAID EpiCoV, cité par ECDC.
.
D’autres variants se propageant rapidement ont également été découverts et sont originaire du Brésil (variant P.1, appartenant au lignage B.1.1.28) et d’Afrique du Sud (variant baptisé 501Y.V2 appartenant au lignage B.1.351).
Marc Gozlan, Royaume-Uni : un variant du coronavirus SARS-CoV-2 porteur de nombreuses mutations. Le Monde. Décembre 2020
Marc Gozlan, Covid-19 : le défi des nouveaux variants. Le Monde. Janvier 2021.
Problématique : Pourquoi ces nouveaux variants se propagent-ils rapidement dans la population ?
A partir de l’exploitation du document 1, proposez une hypothèse à la problématique.
Étape 1 : Identification des mutations de la protéine de surface Spike chez les variants du SARS-CoV-2

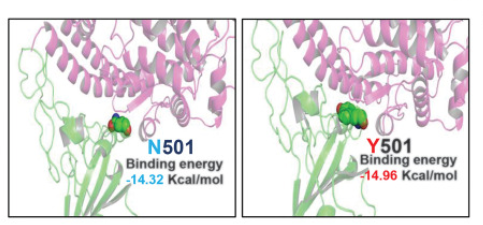
Adaptation of SARS-CoV-2 in BALB/c mice for testing vaccine efficacy. Science. 2020
Étape 2 : pour vérifier votre hypothèse, modélisez à partir du logiciel l’évolution de la population virale selon l’hypothèse proposée.
Aller sur le logiciel en ligne Edu’modèles : https://www.pedagogie.ac-nice.fr/svt/productions/edumodeles/algo/index.htm?expert=0
1- Modifier les paramètres de l’environnement : 50 lignes et 50 colonnes
2- Ajouter des nouveaux agents correspondant aux individus sains immunisés ou non et aux individus infectés par le coronavirus initial ou par un nouveau variant.
Les différents agents :
Sain non immunisé : probabilité de déplacement de 100 %, demi-vie de 0 (infinie), nombre au départ de 200 pour la première modélisation.
Sain immunisé : probabilité de déplacement de 100 %, demi-vie de 0 (infinie), nombre au départ de 0 pour la première modélisation.
Infecté coronavirus initial: probabilité de déplacement de 100 %, demi-vie de 0 (infinie), nombre au départ de 1.
Infecté nouveau variant : probabilité de déplacement de 100 %, demi-vie de 0 (infinie), nombre au départ de 0.
3- Puis ajouter les règles du modèle :
Règle de transmission du coronavirus initial :  Établir un âge minimal (nombre de tours du modèle) de 50 et une probabilité de réaction de 25 % : le contact entre les 2 réactifs « sain non immunisé » et « infecté coronavirus initial » conduira à la production d’un produit « infecté coronavirus initial » dans une probabilité de 25 %. Cocher les cases permettant au produit « infecté coronavirus initial » de se trouver à la même case que le réactif initial « infecté coronavirus initial » et d’hériter de son « âge » puis cocher la case permettant au produit « infecté coronavirus initial » de se retrouver à la place du « sain non immunisé ».
Établir un âge minimal (nombre de tours du modèle) de 50 et une probabilité de réaction de 25 % : le contact entre les 2 réactifs « sain non immunisé » et « infecté coronavirus initial » conduira à la production d’un produit « infecté coronavirus initial » dans une probabilité de 25 %. Cocher les cases permettant au produit « infecté coronavirus initial » de se trouver à la même case que le réactif initial « infecté coronavirus initial » et d’hériter de son « âge » puis cocher la case permettant au produit « infecté coronavirus initial » de se retrouver à la place du « sain non immunisé ».
Procéder de façon comparable pour ajouter la nouvelle règle de transmission du nouveau variant sauf qu’on considère que ce variant se transmet 60 % plus (il faut donc modifier la probabilité de transmission).
Puis ajouter une règle de guérison pour le coronavirus initial: l’individu « infecté coronavirus initial » va donner un individu « sain immunisé » avec une probabilité de 2 % à chaque tour, on ajoute une durée nécessaire à la guérison, soit 700 tours. Ne pas oublier de cocher la case permettant au « sain immunisé » de prendre la même place que l’individu « infecté coronavirus initial ».
On ajoute, avec les mêmes paramètres, une règle de guérison pour le variant du coronavirus.
4- Faire tourner le modèle pendant 1000 tours et observer l’évolution des différents agents sur les courbes obtenues. Puis faites une seconde modélisation en remplaçant l’individu infecté par le coronavirus initial par un individu infecté par un nouveau variant. Et enfin faites une troisième modélisation en mettant un individu infecté par le coronavirus initial et un individu infecté par le nouveau variant. Plusieurs modélisations successives peuvent être faites pour les mêmes paramètres de départ.
5 -Exploiter et comparer les résultats obtenus pour décrire et expliquer la propagation du coronavirus initial et du nouveau variant dans la population.
