L'alignement de séquences et leur comparaison
L'alignement des séquences est utilisé dans deux situations :
- pour comparer des séquences homologues, de longueur comparable,
- pour comparer un gène et l'ARNm qui en est issu pour mettre en évidence la structure morcelée des gènes d'eucaryotes et repérer exons et introns.
La méthode d'alignement multiple, qui est adaptée au premier cas, ne fonctionne, dans le deuxième, que si on ne compare que 2 séquences. Elle fait appel à un logiciel externe spécialisé (ici clustalW2).
Pour comparer un gène et l'ARNm, GénieGen possède une méthode originale qui fonctionne correctement avec plusieurs ARN (cas d'épissage alternatif). C'est l'alignement sur la plus longue séquence.
Pour faciliter l'usage par les élèves, la dernière version de Géniegen offre un menu Action/Alignement qui renvoie directement vers la méthode la plus adaptée aux séquences sélectionnées. Le menu Action/Alignement choisi permet toutefois de choisir une méthode si on le souhaite.
L'alignement multiple
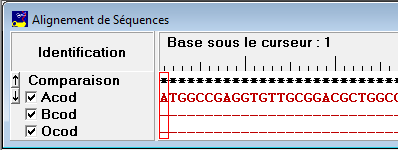
L'alignement multiple est une opération délicate, opérée par des logiciel spécialisés, comme ClustalW2 qui est utilisé par GénieGen. Après alignement, une fenêtre d'alignement s'affiche.
|
|
La première ligne (Comparaison) comporte des étoiles là où toutes les séquences ont la même base (ou le même acide aminé si on compare des protéines). |
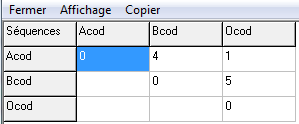
Lorsque la fenêtre d'alignement est sélectionnée, le menu ![]() donne accès à une page qui comporte un tableau de comparaison des séquences.
donne accès à une page qui comporte un tableau de comparaison des séquences.
|
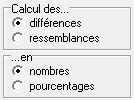
Par défaut, la zone prise en compte pour la comparaison exclut les parties absentes de certaines séquences au début ou à la fin. Par contre les trous "internes" sont comptés comme des différences. Les pourcentages sont donnés par rapport à la longueur des séquences alignées (extrémités exclusives exclues). Ainsi, on évite que le résultat dépende de la séquence de référence. Cela est plus simple pour les élèves. Les choix techniques permettent de modifier ces paramètres. |
dans ce tableau, les valeurs représentent ici le nombre de différences entre chaque couple de séquences. On peut aussi choisir de représenter les ressemblances et exprimer les résultats en pourcentages.
|
L'alignement sur la plus longue séquence
C'est la méthode à utiliser pour la mise en évidence des exons et des introns par comparaison du gène et de l'ARNm (ou des différents ARNm). C'est la méthode directement lancée par le menu Action/Alignement si la plus longue séquence est au moins 1,3 fois plus longue que chacune des autres.
Une première fenêtre demande si on veut conserver les parties identiques (ou aussi différentes s'il n'y a que deux séquences).
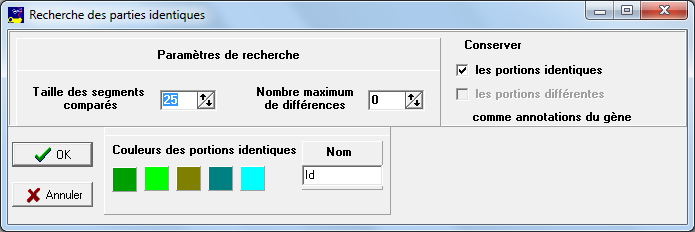
Les carrés de couleur représentent les couleurs qui seront affectées aux parties identiques entre le gène et la première séquence d'ARN (premier carré), la deuxième (deuxième carré)...
Les noms des annotations correspondantes seront Id-A-1 puis Id-A-2... pour les parties identiques entre le gène et la première séquence d'ARN, Id-B-1 puis Id-B-2... pour les parties identiques entre le gène et la deuxième séquence d'ARN... Si on remplace Id par un autre nom dans la case d'édition nom (par exemple Exon) Id sera remplacé par le nom choisi (Exon-A-1...).
Pour changer une couleur, cliquer sur le carré correspondant. et choisir une autre couleur.
Cliquer sur OK pour lancer l'alignement.
La fenêtre d'alignement a ici un intérêt plus limité, dans la mesure où les zones identiques figurent maintenant le long de la séquence d'ADN sous forme de lignes colorées avec les couleurs précédemment choisies.
Voici par exemple la fin du premier Exon commun aux ARN1 et 2 (ils sont ici notés respectivement Id-A-1 et Id-B-2).
 sous les séquences un cadre indique que le curseur se trouve à la fois dans les zones Id-A-1 et Id-B-1 du gène CGRP.
sous les séquences un cadre indique que le curseur se trouve à la fois dans les zones Id-A-1 et Id-B-1 du gène CGRP.
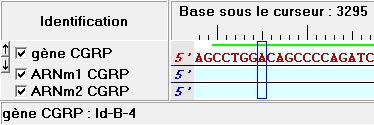
| Plus loin, on trouve un Exon correspondant seulement à l'ARN2(Id-B-4) |
|
| et encore plus loin, on trouve un Exon correspondant seulement à l'ARN1(Id-A-4) | 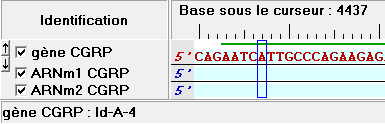 |
Il est possible d'obtenir une représentation graphique de la séquence avec ses exons à l'aide du menu Edition/Dessiner la séquence.
Il faudra alors positionner une à une les étiquettes de légende puis cliquer à côté du cadre délimité par les petits carrés les plus externes pour faire disparaître les marques de positionnement.
Un titre peut être ajouté... L'image obtenue peut être imprimée ou copiée pour être collée dans un compte-rendu. Voir la représentation graphique des séquences.