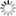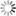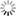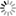Localisation, sur les molécules en 3D, des régions variables des chaînes lourdes et légères
On effectue une coloration sélective des acides aminés 1 à 110 des chaines légères et des acides aminés 1 à 120 des chaines lourdes, sur des molécules 3D d'immunoglobulines, entières (fichier iggtotal.pdb) ou non (fichiers e6o.pdb et nld.pdb). Les atomes des régions colorées sélectivement sont représentés par des sphères rouges dans le cas des chaines lourdes et par des sphères jaunes dans le cas des chaines légères. Le reste de la molécule est représenté en squelette carboné, avec une couleur différente pour chacune des chaînes.
Dans le cas de la coloration effectuée sur une molécule d'immunoglobuline "entière" (fichier iggtotal.pdb), on obtient le résultat suivant :

Dans le cas de la coloration effectuée sur la molécule incomplète correspondant à une branche du "Y" de l'anticorps spécifique d'un motif de la glycoprotéine virale gp41 (fichier 1NLD.pdb) , on obtient le résultat suivant :

Dans le cas de la coloration effectuée sur la molécule incomplète correspondant à une branche du "Y" de l'anticorps spécifique d'un motif de la protéine virale p24 (fichier E60.pdb) , on obtient le résultat suivant :

On s'aperçoit ainsi que, quel que soit l'anticorps étudié, les régions variables des chaines lourdes et légères sont localisées à l'extrémité de chacune des deux branches du Y.