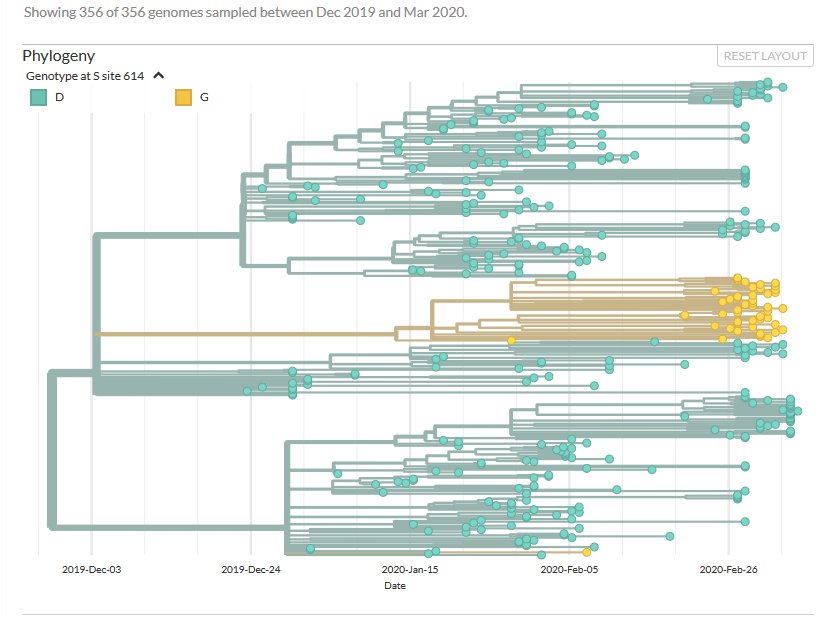
Mise en évidence d’un sous clone dans un arbre phylogénétique
Par Naoum Salamé
—
Dernière modification
16/02/2024 15:13
7 . Mise en évidence d’un sous clone dans un arbre phylogénétique
Il s’agit comme précédemment de l’arbre montrant les relations de parenté entre les 356 génomes viraux séquencés durant les mois de Janvier et de février 2020. Mais cette fois, on a coloré les génomes possédant la mutation A23403G au niveau nucléique ce qui correspond au niveau protéique à la mutation S : D614G
Cliquer sur l'image pour accéder aux données sur Nextstrain.
Exploitation
On peut demander :
- D’indiquer par rapport au génome de la racine de l’arbre les mutations au niveau nucléique et protéique présentes dans le génome du virus de Mexico (C241T ; C3037T ; A23403G ; C14408T ; G28881A ; G28882A ; G28883C ; C13206G au niveau nucléique et S : D614G ; ORF1b : P314L ; N : R203K ; N : G204R et ORF1a A4314G). Cette recherche permet de voir si l’évolution par genèse de sous clones successifs est bien comprise.
- D’indiquer ce que les génomes colorés en jaune de fin février ont en commun avec le génome du virus de Mexico et en quoi ils en diffèrent en se limitant au niveau protéique (Tous ont les mutations S : D614G et P314L ; certains ont en plus les mutations N : R203K et G204R ; aucun n’a la mutation propre au virus de Mexico ORF1a : A4314G).