Contenu des fichiers pour Anagène
Séquences pour Anagène
Origine des données : NCBI (GENBANK)
Avertissement- Dans les banques de données internationales (GENBANK, EMBL, DDBJ) toutes les séquences nucléiques sont en ADN. Exemple : la séquence de référence du Sars-CoV-2. - Les séquences protéiques sont à l'origine en code international 1 lettre. Elles sont transcodées en 3 lettres par Anagène. Exceptions Sars-CoV-2-Référence-ADN-ARN.edi
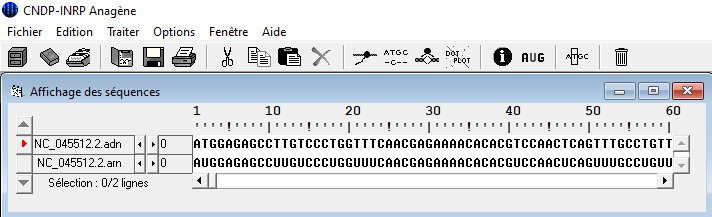
La séquence de référence en ADN et en ARN. : Pour montrer l'équivalence (aux T/U près). Sars-CoV-2-ARN-Référence et complémentaire.edi
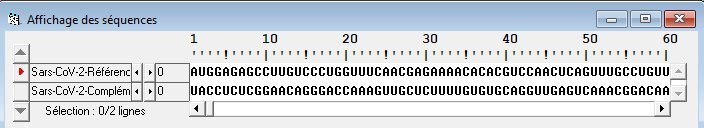
La séquence de référence en ARN mise en parallèle avec sa complémentaire.
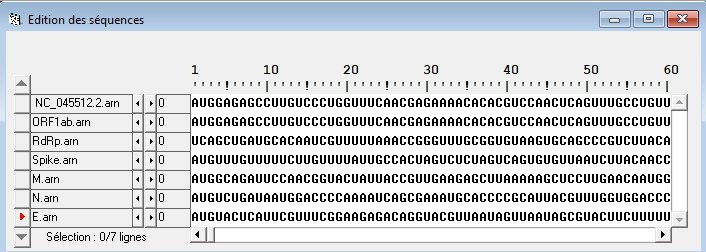
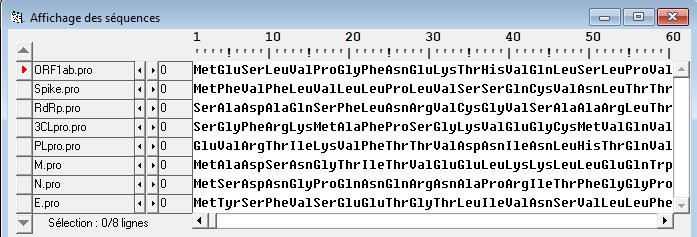
Les principales protéines de Sars-CoV-2. Autres séquences nucléiques (en ADN) ou protéiques
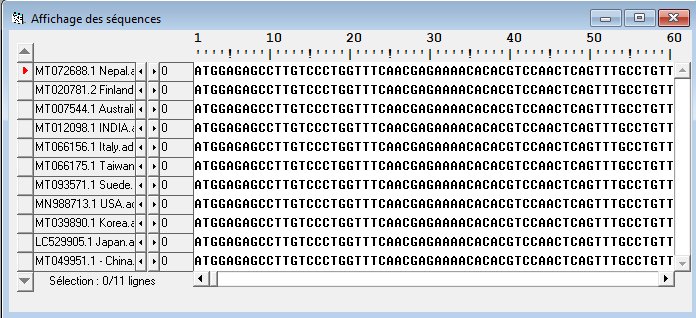
Séquences nucléiques de 12 personnes originaires de Chine. Génomes complets. Comparaison des séquences Les séquences commencent toutes par le premier codon (ATG) de la première protéine et finissent par le codon STOP de la dernière protéine. La comparaison de toutes les séquences avec discontinuités serait longue (plusieurs heures) et risquerait de mettre en défaut le système à cause du volume. A proscrire. On peut utiliser la comparaison simple. Très rapide.
Séquences nucléiques du Sars-CoV-2. 22 Personnes de différentes nationalités. Génomes complets. Comparaison des séquences Les séquences commencent toutes par le premier codon (ATG) de la première protéine et finissent par le codon STOP de la dernière protéine. La comparaison de toutes les séquences avec discontinuités serait longue (plusieurs heures) et risquerait de mettre en défaut le système à cause du volume. A proscrire. On peut utiliser la comparaison simple. Très rapide.
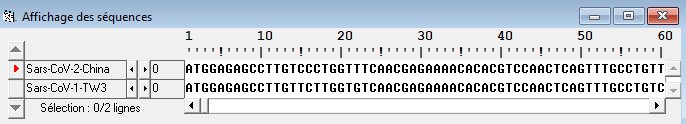
Séquences nucléiques de Sars-CoV-1 et Sars-CoV-2. Génomes complets Comparaison des séquences Les séquences sont longues mais l'alignement avec discontinuités est possible. (temps estimé : 2 minutes). Résultats Cliquer sur le résultat de l'alignement. Cliquer sur l'icone On obtient la valeur absolue et le pourcentage des identités par rapport à la première séquence considérée comme référence. Coronavirus humains-HCoV-bis.edi
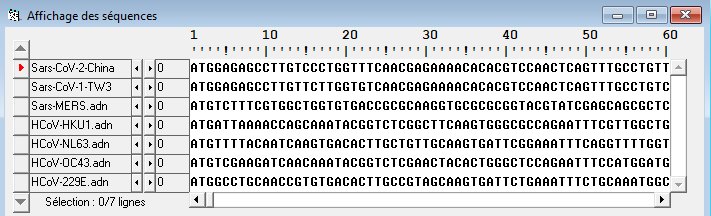
Les séquences de 7 coronavirus humains. Génomes complets. Elles commencent toutes par le premier codon (ATG) de la première protéine et finissent par le codon STOP de la dernière protéine. Comparaison des séquences La comparaison de toutes les séquences avec discontinuités serait longue (plusieurs heures) et risquerait de mettre en défaut le système à cause du volume. A proscrire. On peut utiliser la comparaison avec discontinuités des séquences deux à deux. Patienter (temps estimé 2 minutes). [Ou consulter le fichier aligné, la phylogénie et la matrice des distances avec Phylogène] Résultats Cliquer sur le résultat de l'alignement avec Anagène. Cliquer sur l'icone On obtient la valeurs absolue et le pourcentage des identités par rapport à la première séquence considérée comme référence. Sars-CoV-2 - Surface protéine - Divers individus.edi
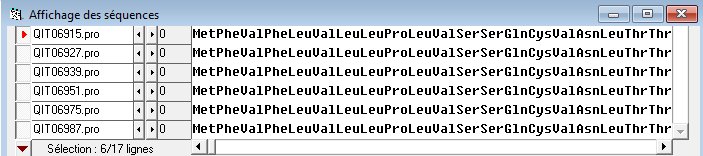
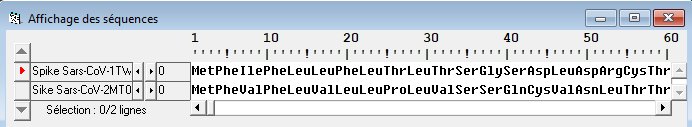
Séquences protéiques "Spike" de 17 individus. Comparaison des séquences Alignement simple ou avec discontinuité. Indifféremment. Résultats Cliquer sur le résultat de l'alignement. Cliquer sur l'icone On obtient la valeur absolue et le pourcentage des identités par rapport à la première séquence considérée comme référence. Spike Sars-CoV2-CoV1-Complets.edi
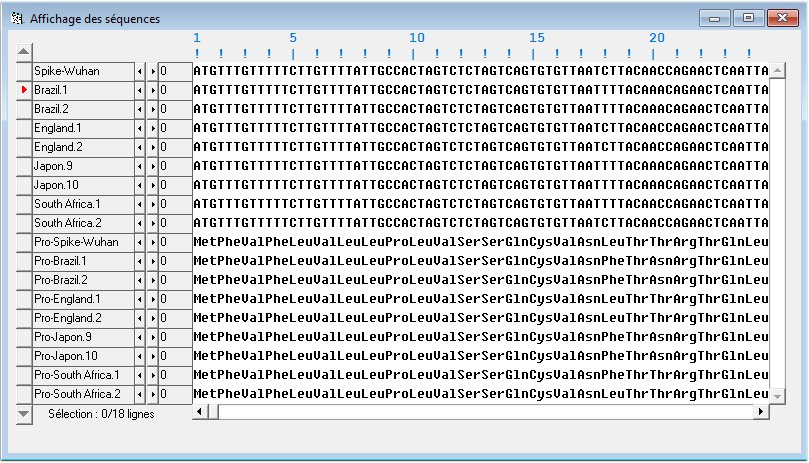
Fichier des "Spike" de Sars-Cov-1 et Sars-CoV-2 Complets. Comporte le domaine de fixation au récepteur ACE2. Séquences protéiques (1265 et 1273 aa) Comparaison des séquences Alignement avec discontinuité, sans problème. Résultats Cliquer sur le résultat de l'alignement. Cliquer sur l'icone On obtient la valeur absolue et le pourcentage des identités par rapport à la première séquence considérée comme référence. Sélection Bre-Eng-Jap-S-Afr.edi Protéine Spike des variants Anglais, Brésilen, Japonais, Sud-Africain du Sars-CoV-2. Au 14-01-2021 Séquences nucléiques et protéiques. 2 représentants de chaque variant.
|