L’origine du Sars-CoV-2 responsable de la pandémie Covid-19
4 : L’origine du Sars-CoV-2 responsable de la pandémie Covid-19
Pour aborder ce sujet, on dispose des documents suivants :
- Séquences des génomes (complets) du Sars-CoV-2, du coronavirus RaTG13 infectant une Chauve-souris rhinolophe, des coronavirus de Chauve-souris : bat_SL_CoV ZC45, et bat_SL_CoV ZXC21, de Pangolin Guangdong.
Fichier pour Anagène : Fichier Sars-divers bat et pangolin.edi
- Phylogeny of Sars-like betacoronavirus including novel coronavirus Sars-CoV-2 Nextstrain
- Séquences nucléiques du gène S de Sars-CoV-2, RaTG13 et du Pangolin. Séquences protéiques S des mêmes coronavirus.
Fichier pour Anagène : Fichier Spike-Sars-CoV2-Sars-Pangolin-Sars-RaTG13-ADN-Pro.edi
- Phylogénies (arbre) de Sars-CoV2, RaTG13 et Pangolin CoV construites à partir de données sur différentes régions du gène S ;
- Séquences du RBD des protéines S du Sars-cov-2, de RaTG13 avec mention des acides aminés cruciaux pour la liaison au récepteur cellulaire ACE2.
Exploitation pédagogique
-
La recherche sur l’origine du Sars-CoV-1 a abouti à l’idée que c’est un coronavirus hébergé par une espèce de Chauve-souris qui était à l’origine du coronavirus humain responsable de l’épidémie 2002-2003. Toutefois la transmission du coronavirus de la Chauve-Souris à l’Homme n’a pas été directe mais a impliqué au moins une espèce intermédiaire la Civette. Dès l’élucidation du génome du Sars-CoV-2 en janvier 2020, des chercheurs se sont attaqués au problème de l’origine de ce virus humain émergent et, se basant sur les connaissances acquises à propos du Sars-CoV-1, ils ont cherché des coronavirus de Chauve-souris qui pourraient être à l’origine du nouveau virus humain. Ils ont ainsi séquencé le génome de 3 coronavirus : RaTG13, SlZc45 et SLZXC21 présents chez des Chauve-souris. D’autres chercheurs dont l’attention a été attirée par des informations suivant lesquelles des pangolins manifestaient des symptômes proches de ceux du Covid-19 humain, ont trouvé des coronavirus chez ces animaux malades et séquencé les génomes de ces virus.
-
La comparaison avec Anagène des séquences des génomes des 3 coronavirus de chauve-souris et de celui trouvé chez les pangolins avec le génome de Sars-CoV-2 permet donc de discuter si ces coronavirus animaux peuvent être à l’origine du virus humain. On constate que c’est le génome du RaTG13 qui a la plus forte similitude – (plus de 96% d’identité) avec le Sars-CoV-2 ce qui est un fort argument pour dire qu’il peut être à l’origine du Sars-CoV-2. Le virus du Pangolin a aussi une forte similitude avec le virus humain de l’ordre de 90%. Les coronavirus ZC45 et ZXC21 de Chauve-souris ont une plus faible identité avec le Sars-CoV-2.
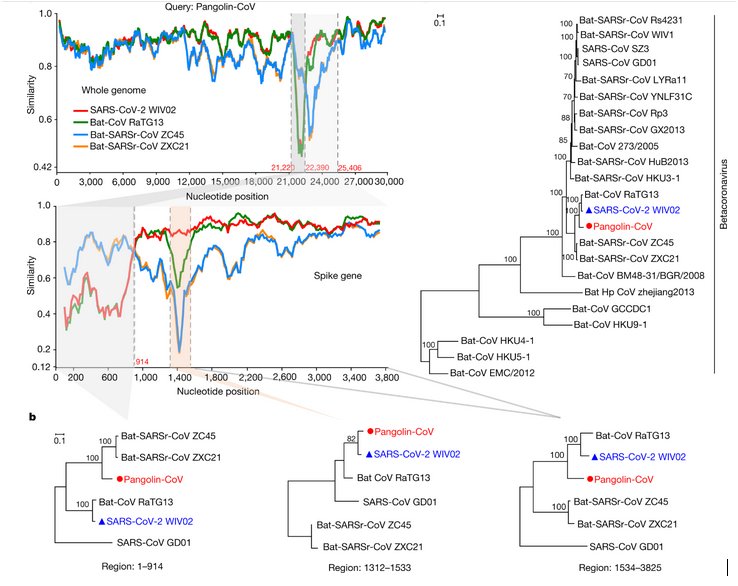
Cette comparaison peut se prolonger par la réalisation d’un arbre phylogénétique simple relatif à ces virus qu’on peut confronter avec l’arbre beaucoup plus complet fourni par le site « Nextstrain ». (Phylogeny of sars-like betacoronavirus including novel coronavirus Sars-CoV-2.) -
Une analyse plus fine du gène qui code pour la protéine S qui permet la fixation du coronavirus à des récepteurs cellulaires (ACE2 pour le sars-CoV-2 ) des cellules de l’animal infecté, fournit des informations sur une modalité de l’évolution des génomes des coronavirus. Pour cela il faut comparer avec Anagène les séquences nucléotidiques des gènes S dans trois régions différentes : de 1 à 914 nucléotides, de 1312 à 1533 nucléotides et de 1534 à 3825 nucléotides.
-
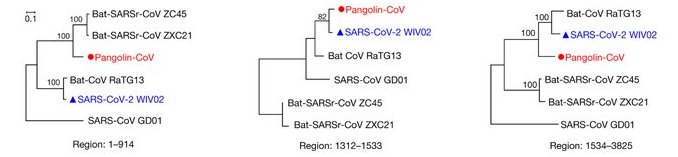
On constate que le génome du pangolin est plus similaire à ceux des coronavirus ZXC21 ZC45 qu’à ceux du RaTG13 et du Sars-CoV-2 dans la première partie (1-914) du gène. En revanche dans la région 1312-1533 la séquence du coronavirus du Pangolin est plus proche de celle du Sars-CoV-2 que des coronavirus de Chauve-souris, RaTG13 compris. Enfin dans la dernière partie 1533- 3825, on retrouve la même phylogénie que celle fournie par la comparaison des génomes globaux. Cela est traduit par les 3 arbres phylogénétiques ci-dessous.

-
Ainsi le gène S du coronavirus du Pangolin, possède une région qui semble provenir du coronavirus ZC45 ou ZXC21 et une région ayant pour origine le RaTG13. Cela traduit une recombinaison génétique associant des fragments de deux génomes différents. Cela est possible si deux virus différents infectent les mêmes cellules et si cela est suivi d’un phénomène ayant lieu lors de la réplication des génomes viraux dans la cellule parasitée.
Ainsi l’évolution du génome des coronavirus peut se faire par mutation et par recombinaison génétique ; cette dernière modalité a des effets plus drastiques que la mutation. -
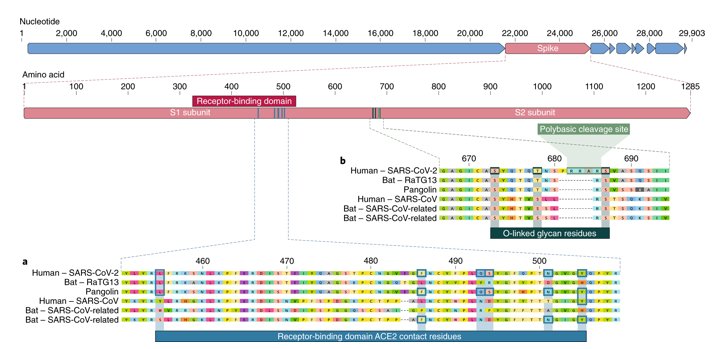
La comparaison du gène de la protéine S indique que dans la région 1312-1533, la séquence du Pangolin a une identité nettement plus forte avec celle du Sars-CoV-2 que ne l’a la séquence du RaTG13. Or cette région correspond au RBD, c’est-à-dire à une région du gène S qui code pour le domaine de fixation au récepteur ACE2 des cellules humaines parasitées. Cela indique que le coronavirus RaTG13 malgré la très grande identité globale de son génome avec celui de Sars-CoV-2 ne peut être le virus qui, directement transmis à l’homme, a été à l’origine de Sars-CoV-2. Cela est confirmé par une analyse fine de ce domaine RBD. On a identifié 6 acides aminés du RBD de la protéine S du Sars-CoV-2 qui jouent un rôle crucial dans la liaison au récepteur ACE2 : L455, F486, Q493, S494, N501 et Y 505.
Le document ci-dessous renseigne sur les séquences des RBD des coronavirus précédemment envisagés.

-
On peut constater que le RBD de RaTG13 n’a qu’ un seul des 6 acides aminés contrairement au coronavirus du pangolin qui en possède les 6.
Cela indique que le coronavirus RaTG13 de la Chauve-souris Rhinolophus affinis ne peut être le virus qui a réussi à franchir la barrière d’espèce et causé la pandémie du Covid-19
