Les phénotypes groupes sanguins
Les phénotypes groupes sanguins
La découverte des groupes sanguins
C'est Richard Lower, médecin britannique, qui effectua le premier, en 1665, des transfusions sanguines d'animal à animal. Mais le premier à transfuser avec succès du sang d'animal, un agneau, à un jeune garçon anémique de 16 ans, deux ans plus tard, fut Jean Baptiste Denis.
Cependant, les incidents se multiplièrent et, si la transfusion réussissait parfois, elle ne faisait souvent que précipiter le décès (l'agglutination des hématies du donneur suivie d'une lyse de ces hématies entraînait des troubles métaboliques importants, la destruction des reins...). Bientôt, les transfusions sanguines furent interdites et n'allaient être reprises que vers la fin du siècle dernier.
Lorsque Karl Landsteiner (1868-1943) commença ses recherches comme assistant à l'Université viennoise de l'hygiène, il se proposa de surmonter ces difficultés. Il avait constaté que, lorsque l'on mélangeait du sang de plusieurs personnes, on observait parfois une agglutination des hématies et parfois non. Il rassembla alors des échantillons de sang de ses collègues et les mélangea deux par deux. C'est ainsi qu'il découvrit l'existence des groupes sanguins, qu'il nomma A, B et O, et il fit connaître les résultats de ses recherches en 1900. Le quatrième groupe sanguin (AB) ne fut découvert que l'année suivante. Cette découverte permis d'établir une méthode de transfusion sanguine sûre.
En reconnaissance pour ses travaux, Landsteiner fut nommé professeur de pathologie à l'Université de Vienne, mais il ne s'y plut pas et partit pour les Pays-Bas puis pour l'Institut Rockfeller de New-York. Il découvrit plus tard les groupes sanguins M et N, ainsi que le facteur rhésus, en collaboration avec Phillip Levine. Lansdsteiner reçut le Prix Nobel de médecine en 1930.
Les marqueurs des groupes sanguins principaux
Les marqueurs des groupes sanguins principaux sont des glycolipides présents dans la membrane des hématies. Il existe deux types de marqueurs : des marqueurs de type A et des marqueurs de type B.
Ces marqueurs déterminent les groupes sanguins principaux :
- présence de marqueurs A seulement : groupe sanguin A ;
- présence de marqueurs B seulement : groupe sanguin B ;
- présence de marqueurs A et de marqueurs B : groupe sanguin AB ;
- absence de marqueurs A et B : groupe sanguin O .
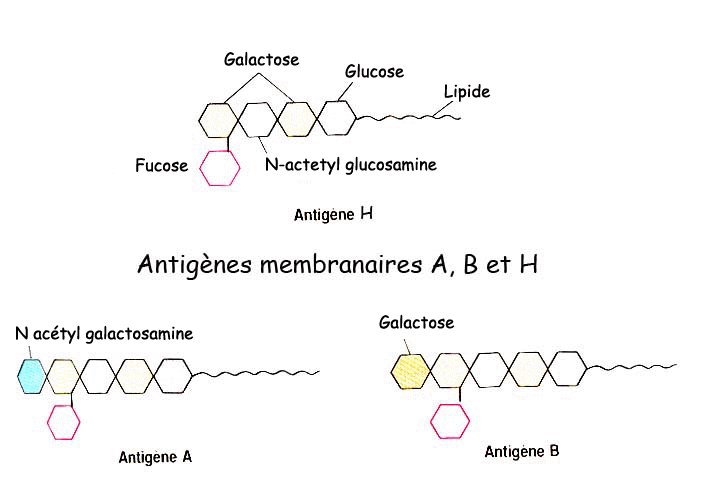
Les deux dernières étapes de la chaîne de biosynthèse des marqueurs A et B
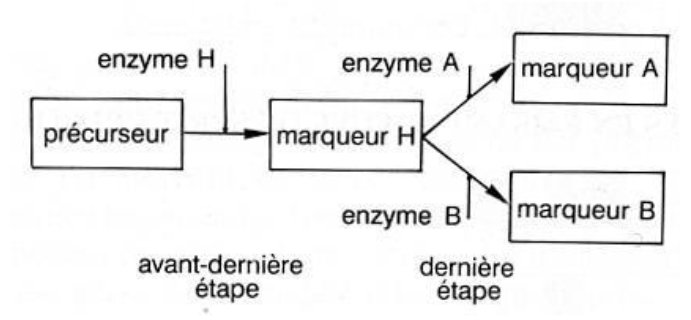
Le gène Fut1 code pour la synthèse de l'enzyme H (EH). Le gène des groupes sanguins principaux code pour la synthèse des enzymes A et B (EA, EB).
Les phénotypes sanguins A, B et O
Ils sont définis respectivement par la possession des antigènes membranaires A, B et H. Cependant il est possible de retrouver les divers niveaux d’expression du phénotype vus avec les hémoglobinopathies :
- phénotype macroscopique : il ne se révèle pas spontanément mais il se manifeste si on met les globules rouges d’un individu en contact avec des sérums tests anti A et anti B et aussi en cas de transfusion incompatible ;
- phénotype cellulaire : c’est la panoplie des marqueurs relatifs à ce système de groupe sanguin présents dans la membrane des hématies mais aussi à la surface des autres cellules de l’organisme ;
- phénotype moléculaire : les marqueurs membranaires résultent de l’action de l’enzyme produit de l’expression du gène ; cette enzyme est en somme le phénotype moléculaire primaire.
Les séquences
ABO ADN.edi : séquences nucléiques codantes des allèles A101, B101 et O01 ;
ABO Protéines.edi : séquences protéiques des enzymes A 101, B101 et O01 ;
ABO Génotypes Famille1.edi : séquences nucléiques codantes des allèles du gène ABO de chacun des membres de la famille 1 (génotype de Paul : A//O, de
Lisette : B//O, de Martine : O//O, de Luc : A//B, de Marion : A//O).
ABO Génotypes Famille2.edi : séquences nucléiques codantes des allèles du gène ABO de chacun des membres de la famille 2 (génotype de Paul : A//B, de Pascale : O//O, de Roland : B//O, de Magali : A//O, de Bérénice : B//O) ;
Fut1 ADN.edi : séquences codantes des allèles de référence du gène Fut1 (l’allèle Fut1H code pour une enzyme H fonctionnelle ; l’allèle Fut1h1 code pour une enzyme h non fonctionnelle) ;
Fut1 Protéines.edi : séquences protéiques des enzymes H (fonctionnelle) et h1 (non fonctionnelle) ;
ABO Polymorphisme.edi : séquences nucléiques codantes de dix allèles du gène ABO (allèles A101, A102, A103, A201, B101, B102, B103, O01, O04, O06) ; tous les allèles A et B fournis codent pour des enzymes A et B fonctionnelles.
Fut1 Polymorphisme.edi : séquences nucléiques codantes de 6 allèles du gène Fut1 (allèles fut1, codant pour une enzyme H fonctionnelle, allèles Fut1h1 à Fut1h5, codant pour des enzymes h non fonctionnelles) ;
Fut1 Génotypes famille2.edi : séquences nucléiques codantes des allèles du gène Fut1 de chacun des membres de la famille 2 (génotype de Paul : h1//h1, de Pascale : H1//h1, de Roland : h1//h1, de Magali : H1//h1, de Bérénice : H1//h1).
Pistes d’exploitation pédagogiques des données fournies
L’analyse des documents et des données concernant le phénotype groupe sanguin permet d’aborder les notions suivantes :
- le phénotype peut se définir à différents niveaux, et le phénotype moléculaire détermine le phénotype cellulaire et macroscopique ;
- le phénotype moléculaire s’explique par le génotype ;
- un même phénotype peut correspondre à plusieurs génotypes ;
- plusieurs gènes peuvent être impliqués dans la réalisation d’un phénotype.
La diversité des phénotypes et les relations génotype/phénotype (chez les homozygotes)
- Le phénotype clinique est mis en évidence par l'observation des accidents de transfusion ;
- le phénotype cellulaire par les marqueurs membranaires ;
- le phénotype moléculaire par l'enzyme codée par le gène ABO (enzyme sous la forme A, B ou inactive O).
Remarques :
- il est important pour la suite de la démarche que l’élève ait bien compris que le groupe O est lié à l’absence de marqueurs A et B ;
- qu'il comprenne également que le phénotype moléculaire ne peut être le marqueur membranaire (qui n’est pas une protéine) mais l’enzyme permettant sa synthèse.
La comparaison des séquences des différentes protéines ABO et la mise en relation avec les phénotypes cliniques correspondants permet de discuter des relations entre phénotype moléculaire, cellulaire et clinique. Les connaissances sur les protéines enzymatiques seront mobilisées pour expliquer les observations. On montrera ainsi que les phénotypes alternatifs sont dus à des différences dans les protéines concernées.
Remarque pratique :
La mise en relation des différences observées entre les allèles avec celles observées entre les polypeptides est facilitée si l’on demande des comparaisons simples dans les deux cas.
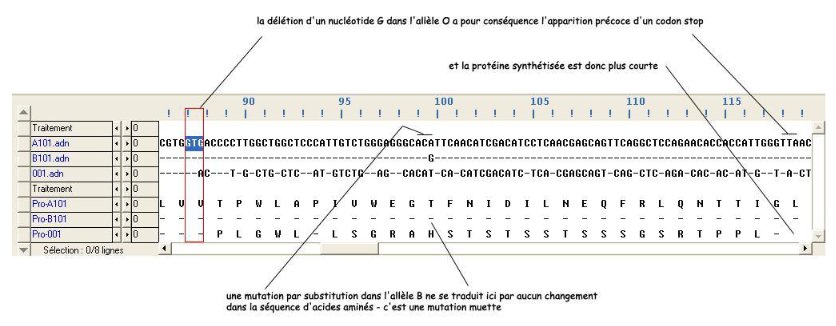
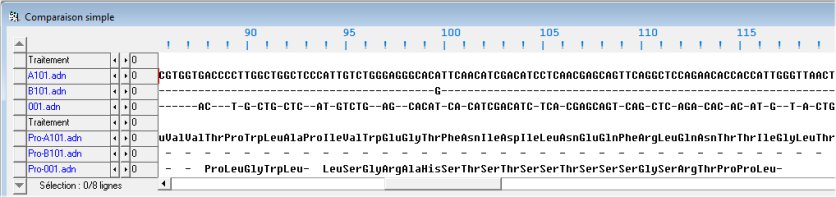
Bilan
Les enzymes EA et EB diffèrent par 4 acides aminés, en position 174, 233, 264 et 266. Ces différences sont à mettre en relation avec les différences d'activité de ces enzymes (elles ne catalysent pas la même réaction) : certains des acides aminés différents doivent se situer au niveau du site actif de l'enzyme.
L'"enzyme" O est beaucoup plus courte que les deux autres (115 a.a. seulement). Cette molécule protéique ne doit donc pas pouvoir prendre une forme spatiale correcte, ce qui explique son inactivité.
La mise en relation des différences observées au niveau des quelques allèles sélectionnés ici avec les différences observées au niveau protéique permet de discuter de l’effet des mutations.
- l’allèle O diffère de l’allèle A par une délétion d’un nucléotide en position 261, ce qui entraîne un décalage du cadre de lecture et donc une modification précoce de la séquence d’acides aminés. Cela entraîne aussi l’apparition précoce d’un codon stop, donc une chaîne polypeptidique plus courte (116 acides aminés au lieu de 353) ;
- L’allèle B diffère de l’allèle A par 7 nucléotides ; il s’agit à chaque fois de mutations par substitution. Trois de ces mutations sont des mutations muettes (positions 297, 667 et 930) ; les quatre autres sont des mutations faux-sens (positions : 526, 703, 803).
Le gène ABO est en fait très polymorphe. Le fichier ABO Polymorphisme.edi fournit les séquences de plusieurs autres allèles de ce gène.
Un travail similaire à celui qui précède peut être fait à partir de ces allèles et des séquences protéiques correspondantes.
La complexité des relations gènes/phénotype
La mise en évidence de la complexité des relations génotype/phénotype peut être abordée à partir de l’exemple des deux familles proposées.
Pour chaque famille, sont mises à disposition les données suivantes :
- un arbre qui n’est là que pour préciser le phénotype des membres d’une famille donnée (et non pour initier un raisonnement en fonction des lois de l’hérédité) ;
- les séquences des deux allèles du gène ABO que possèdent tous les membres de la famille ;
- les séquences des allèles ABO considérés comme références .
Étude de la famille 1
L’étude de cette famille permet de se familiariser avec la méthode de détermination des génotypes et de discuter des relations de dominance/récessivité.

O, A, B : groupes sanguins déterminés à l’aide de sérums tests révélateurs des marqueurs A et B.
La comparaison des allèles de chaque individu avec les allèles de référence du gène ABO permet de déterminer le génotype de chacun des membres de la famille : le génotype de Paul est A//O, celui de Lisette est B//O, celui de Martine est O//O, celui de Luc est A//B et celui de Marion est A//O.

La mise en relation du génotype et de phénotype pour les individus hétérozygotes permet ensuite de discuter des relations de dominance/récessivité (ces relations ne peuvent se définir qu’à l’échelle du phénotype clinique et non à l’échelle moléculaire ; en effet, les deux allèles s’expriment dans la cellule).
Étude de la famille 2
L’étude de cette famille permet de découvrir un aspect de la complexité des relations génotype/phénotype : un phénotype peut dépendre de l’expression de plusieurs gènes. Il s’agit de mettre l’élève en situation de découverte du problème.
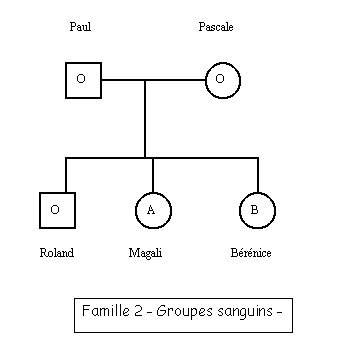
La comparaison des allèles du gène ABO de chaque individu avec les allèles de référence de ce gène permet de déterminer le génotype de chacun : le génotype de Paul est A//B et celui de Magali est A//O, celui de Pascale est O//O, et celui de Roland et de Bérénice est B//O.
Si l’on s’en tenait au fait que le phénotype groupe sanguin ne dépend que du seul gène ABO, et que l’allèle O est récessif par rapport à A et B, il y a une incohérence pour Paul et Roland ; possédant un allèle A ou B, ils ne devraient pas être de groupe O. Il faut donc imaginer que le phénotype groupe sanguin ne dépend pas que du gène ABO.
Des renseignements complémentaires sont alors fournis concernant la synthèse des marqueurs A et B : cette synthèse ne résulte pas d’une seule réaction, mais d’une chaîne de biosynthèse. Cette chaîne nécessite plusieurs réactions chimiques catalysées chacune par une enzyme différente, codée chacune par un gène différent. Il peut donc y avoir plusieurs origines au groupe sanguin O : inactivité de l’enzyme O, mais aussi inactivité de l’enzyme H (codée par le gène Fut1), donc non synthèse du marqueur H.
Remarque
Un travail peut être effectué sur les allèles du gène Fut1 et les protéines correspondantes pour discuter, comme il a été fait pour le gène ABO, des relations entre génotype/phénotype moléculaire et phénotype clinique. Les mutations présentes dans les allèles du gène Fut1 entraînent des modifications de la séquence d’acides aminés de l’enzyme h, responsables de l’inactivité de l’enzyme et d’un phénotype groupe sanguin O.
Résultats de comparaisons simples entre les allèles du gène Fut1 d’une part, et entre les enzymes H correspondantes d’autre part :
- Allèle Fut1h1 : diffère de l’allèle Fut1cod (allèle normal, de référence) par une substitution en position 695 – le remplacement d’un nucléotide G par un A transforme le codon TGG en codon stop TAG. Le polypeptide synthétisé est donc plus court (230 acides aminés au lieu de 399) et donc non fonctionnel.
- Allèle Fut1h2 : diffère de l’allèle Fut1cod par une délétion d’un nucléotide en position 990 ; il s’ensuit un décalage du cadre de lecture et l’apparition précoce d’un codon stop. Le polypeptide synthétisé est donc plus court (334 acides aminés au lieu de 399) et non fonctionnel.
- Allèle Fut1h3 : diffère de l’allèle Fut1cod par une substitution en position 721 (T/C). Il s’agit d’une mutation faux-sens qui entraîne le changement d’un acide aminé dans la séquence du polypeptide. Cette différence suffit à rendre l’enzyme non fonctionnelle.
- Allèle Fut1h4 : diffère de l’allèle Fut1cod par une substitution en position 442 (G/T). Il s’agit d’une mutation faux-sens qui entraîne le changement d’un acide aminé dans la séquence du polypeptide. Cette différence suffit à rendre l’enzyme non fonctionnelle.
- Allèle Fut1h5 : diffère de l’allèle Fut1cod par deux substitutions en position 460 (T/C) et 1042 (G/A). Il s’agit de mutations faux-sens qui entraînent le changement de deux acides aminés dans la séquence du polypeptide. Ces différences suffisent à rendre l’enzyme non fonctionnelle.
La comparaison des allèles du gène Fut1 des membres de la famille 2 avec les allèles Fut1 de référence (Fut1 ADN.edi et Fut1 Génotypes famille2.edi) permet de déterminer leur génotype : Pascale, Magali et Bérénice sont de génotype H1//h1 ; Paul et Roland sont de génotype h1//h1. Ainsi, l’incohérence constatée auparavant pour Paul et Roland trouve ici une explication : bien que possédant un allèle A ou B, ils sont de groupe O car la synthèse du précurseur H ne peut avoir lieu, l’enzyme h1 étant inactive.
On montre ainsi qu’un phénotype peut dépendre de plusieurs gènes et qu’un même phénotype clinique peut correspondre à plusieurs génotypes.