Graphique de ressemblance (DotPlot)
Généralités
Schématiquement, la comparaison de deux séquences a pour objectif de mettre en évidence des ressemblances/différences globales ou locales.
Généralement, la comparaison avec discontinuités (avec les algorithmes de CLUSTAL intégrés à Anagène et à Phylogène) est appropriée pour bien situer les ressemblances et différences locales. Elle est moins pertinente pour montrer d'un seul coup d'oeil les similitudes globales, surtout lorsque les séquences sont longues (on ne voit pas toute la comparaison, et l'algorithme est déficient quand les deux séquences sont très différentes ou de longueurs disproportionnées).
Le DotPlot est une des techniques de comparaison les plus anciennes mais qui reste très efficace pour les comparaisons globales. Elle peut s'appliquer à des séquences différentes ou identiques (dans ce cas, c'est l'analyse des éventuelles répétitions à l'intérieur de la séquence qui est visée).
On peut consulter sur ce point plusieurs sites notamment ceux qui font du DotPlot en ligne :
http://www.vivo.colostate.edu/molkit/dnadot/index.html
http://www.vivo.colostate.edu/molkit/dnadot/bkg.html
et
http://mobyle.pasteur.fr/cgi-bin/portal.py?#forms::polydot
1 - Le principe de la comparaison
La comparaison ne peut avoir lieu qu'entre séquences de même type : ADN, ARN ou polypeptide.
Il s'agit de placer l'une des séquences horizontalement, l'autre verticalement formant ainsi une matrice. Si les deux séquences sont de longueurs différentes, la plus longue est toujours placée horizontalement (en haut).
Supposons que l'on compare une courte séquences d'ARN avec elle-même avec la fonction DotPlot ![]() d'Anagène 2.
d'Anagène 2.
a - Le traitement par défaut (avec les paramètres du logiciel)
On obtient le tracé suivant :
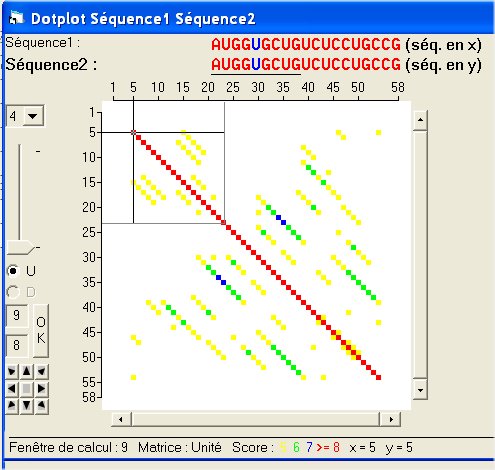
Par défaut le logiciel compare les deux séquences par groupes de 9 éléments (Fenêtre glissante).
Il admet que deux groupes comparés sont identiques si au moins 8 éléments sur 9 sont identiques ![]() (tolérance de 1).
(tolérance de 1).
En outre, toujours par défaut, il affiche 4 niveaux de similitude ![]() correspondant à :
correspondant à :
- point jaune : 5 éléments identiques sur 9
- point vert : 6 éléments identiques sur 9
- point bleu : 7 éléments identiques sur 9
- point rouge : 8 ou 9 éléments identiques sur 9
Signification de la diagonale rouge
Le premier point rouge tracé traduit la comparaison des 9 premiers éléments de chacune des séquences, de la position 1 à la position 9, le point étant placé au milieu du segment (donc en position 5).
Le deuxième point rouge correspond à la comparaison dans les deux séquences des 9 éléments de la position 2 à la position 10, et ainsi de suite.
La diagonale rouge traduit donc l'identité des deux séquences sur toute leur longueur.
Signification des autres petites diagonales
Que signifient les points en dehors de la diagonale rouge et notamment ceux qui forment de petites diagonales ? Elles indiquent un certain degré de similitude entre une portion de la séquence horizontale et une autre de la séquence verticale (dans notre exemple, entre deux régions de la même séquence). Sur le graphique suivant, une de ces petites diagonales est bordée d'un filet magenta).

Ces similitudes sont compréhensibles étant donné qu'il n'y a que 4 types de nucléotides.
Si ces petites diagonales étaient en rouge, elles indiqueraient des segments identiques (à la tolérance près), donc des répétitions à l'intérieur de la séquence.
b - Ajustement des valeurs par défaut
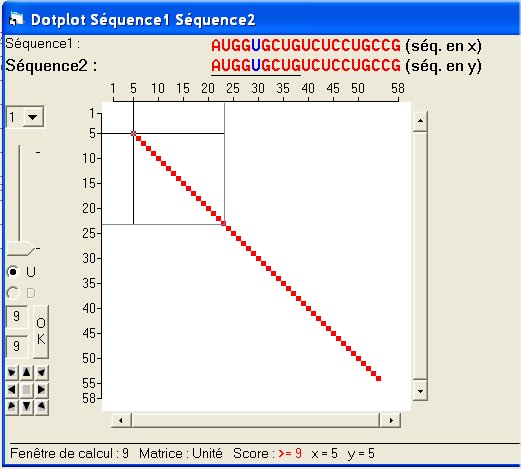
Considérons le cas où on recherche les très fortes similitudes entre deux séquences. On fixe la contrainte maximum (9 éléments identiques sur 9) et on demande d'afficher un seul niveau de similitude qui correspond à la contrainte.
Dans ce cas, on obtient pour cette comparaison le résultat suivant :
2 - Utilisation pratique du DotPlot dans Anagène
- Valeurs par défaut
| Niveau de similitude | |
| Fenêtre glissante de 9 avec une tolérance de 1 | |
 |
Index positionné sur le premier point tracé. Les nucléotides précis correspondant à sa position sont figurés en bleu sur la séquence, au milieu de la fenêtre glissante. |
 |
Bloc d'orientation pour déplacer l'index sur le graphique. La position de l'index en x et en y est indiquée à tout moment en bas du graphique. |
Ces valeurs sont à ajuster en fonction de l'objectif recherché à travers la comparaison. Choisir toujours une valeur impaire pour la fenêtre de comparaison.
- Comparaisons multiples
La comparaison de plusieurs graphiques de ressemblance peut être utile. Opter dès le début pour une visualisation des fenêtres en mosaïque.

