Cycle du virus de la grippe aviaire
VIDEO: animation montrant le cycle intra-cellulaire du virus de la grippe
Source: "La grippe en face" , Edition Xavier Montauban, avec l'aimable autorisation de l'éditeur
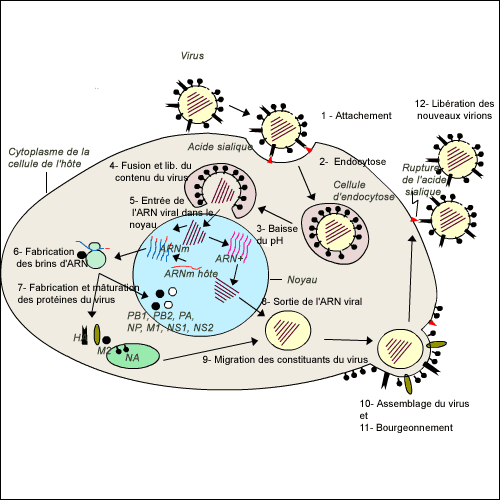 |
Schéma du cycle viral des Orthomyxoviridae (type H5N1) dans une cellule de Mammifère Source |
Le cycle viral des Orthomyxoviridae peut se décomposer en 6 étapes:
1. Attachement et endocytose ( phases 1 et 2)
Pour devenir infectieux le virus doit être mis en présence d'une protéase spécifique du tractus respiratoire qui va cliver l'hémagglutinine en HA1 et HA2, à ce moment là l'hémagglutinine reconnaît les acides sialiques présents à la surface de la cellule hôte ce qui entraîne l'attachement du virus à la surface cellulaire. Un processus d'endocytose a lieu , le virus se trouve alors enfermé dans un endosome.
2. Libération des segments ribonucléoproteiques ( phases 3 et 4)
Le pH chute dans l'endosome à une valeur de 5 ce qui va déclencher un changement de conformation de l'hémagglutinine. La partie HA2 va former une structure amphiphile dans la membrane de l'endosome ce qui qui va favoriser la fusion des membranes de l'endosome et de la membrane qui enveloppe la particule virale, il y a alors libération des segments ribonucléoproteiques dans le cytosol.
3. Migration des huit segments d'ARN vers le noyau ( phase 5)
La migration des segments ribonucléoprotéiques est possible grâce à la protéine NP qui recouvre les ARN viraux, puis les ARN passent par les pores nucléaires de la cellule hôte.
4. Transcription et traduction ( phases 6 et 7)
Dès leur arrivée dans le noyau les ARNs viraux à polarité négative sont transcrits en ARNs à polarité positive. La synthèse des ARNm est assurée par les complexes moléculaires basiques formés des protéines PB1 et PB2 et par le complexe moléculaire acide PA, ils jouent le rôle des ARNpolymérase-ARN dépendante. Ces ARNms sont complétés d'une queue polyA, ainsi maturés, ils sont exportés dans le cytoplasme pour être traduits. Les ARNs positifs non polyadénylés restent dans le noyau et servent de matrice pour la synthèse d'ARNs négatifs génomiques.
Les proteines virales NP, NS2, M1, PA, PB1 et PB2 synthétisées dans le cytoplasme de la cellule hôte sont acheminées vers le noyau. Les protéines de l'enveloppe HA, NA et M2 sont exportées vers la membrane de la cellule hôte. Les ARNs négatifs du génome viral s'associent avec la protéine NP pour former les ribonucléoprotéines associées au complexe polymérase.
5. Encapsidation et bourgeonnement ( phases 8, 9, 10 et 11)
Afin que le virus s'assemble il faut que les protéines virales et les segments ribonucléoproteiques soient présents au niveau de la membrane. Les protéines HA et NA vont s'insérer grâce à un grand remaniement dans la membrane de la cellule hôte. Sur la face interne de la membrane cytoplasmique, les protéines M1 et M2 vont former la matrice. C'est au niveau de cette matrice que viennent s'assembler les nucléocapsides et les protéines virales de l'enveloppe par un processus de bourgeonnement. Les ARNs négatifs du génome viral s'associent avec la protéine NP pour former les ribonucléoprotéines associées au complexe polymérase. C'est surtout la protéine M1 qui est importante pour l'encapsidation et le bourgeonnement.
6. Libération des nouveaux virions
Les nouveaux virions formés par bourgeonnement, se détachent de la cellule par clivage de l'hémagglutinine par la neuraminidase. Une fois libérées les particules virales diffusent à travers la couche de mucus et vont infecter d'autres cellules de l'épithélium respiratoire. La cellule hôte épuisée par la réplication virale devient alors la cible de la réponse immunitaire cytotoxique et est progressivement détruite.
