Le chargement et l'affichage des séquences
Retour à la présentation du logiciel
Pour commencer un travail avec une série de séquences, utiliser le menu Fichier/Charger des séquences. Cela efface définitivement toutes les séquences qui auraient été préalablement chargées.
Pour ajouter des séquences à celles qui sont déjà affichées, on peut opérer à partir d'un fichier avec le menu Fichier/Ajouter des séquences ou entrer manuellement une ou des séquences (menu Fichier/Entrée d'une séquence).
Après leur chargement, les séquences sont affichées suivant leur nature. Suivant les cas, l'ADN peut être représenté par un brin ou les deux (comme dans l'exemple ci-dessous).

Si l'ADN est représenté par ses 2 brins, ils sont référencés sous le même nom. S'il y a à la fois des séquences d'ADN (ou d'ARN) et de protéines, l'affichage des protéines se fait avec le code à 3 lettres. Un curseur bleu encadre alors un acide aminé, et le cas échéant, le triplet de bases situé au-dessus. La position du curseur dans les différentes séquences est clairement indiquée.
Pour déplacer le curseur, on peut utiliser les flèches du clavier ou cliquer sur la séquence. Un curseur horizontal permet d'explorer la séquence dans son ensemble : 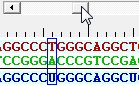
Si la séquence d'ARN et la protéine sont calculées par le logiciel à partir de la séquence d'ADN, les séquences auront le même titre.
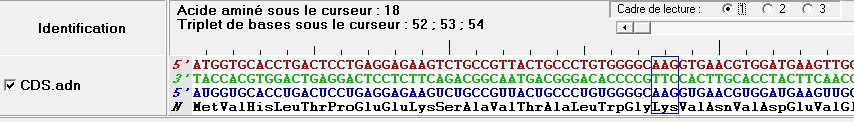
Dans ce cas, l'utilisateur peut choisir le cadre de lecture pour la traduction.
Si on compare deux ou plusieurs séquences 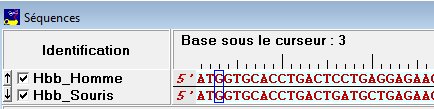 on se facilitera la tâche en utilisant le mode "comparaison" (menu Affichage/Comparaison)
on se facilitera la tâche en utilisant le mode "comparaison" (menu Affichage/Comparaison)
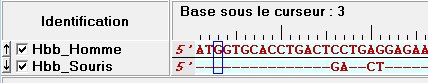 La séquence de la première ligne sert alors de séquence de référence.
La séquence de la première ligne sert alors de séquence de référence.
Les séquences d'ADN peuvent être affichées sous la forme d'un ou deux brins d'ADN, leur transcription en ARN ou leur traduction en protéine peuvent aussi être demandées.
Les séquences d'ARN peuvent éventuellement aussi être associées à leur traduction en protéine.
Les séquences de protéines sont affichées avec le code à 3 lettres. S'il n'y a pas de séquence d'une autre nature, l'affichage en code 1 lettre est possible (menu Affichage/Code 1 lettre - recliquer sur le même item pour revenir au code 3 lettres).
Les choix d'affichage peuvent se faire de trois manières : globalement, par séquences ou par catégorie, à partir du menu Affichage/Choix d'affichage.
1- Choix d'affichage globaux
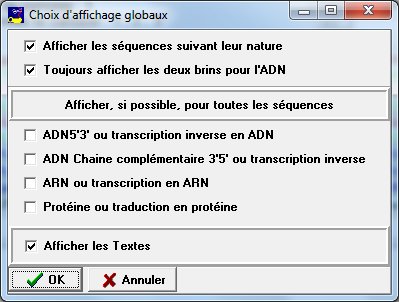 |
|
2 - Choix d'affichage par séquence
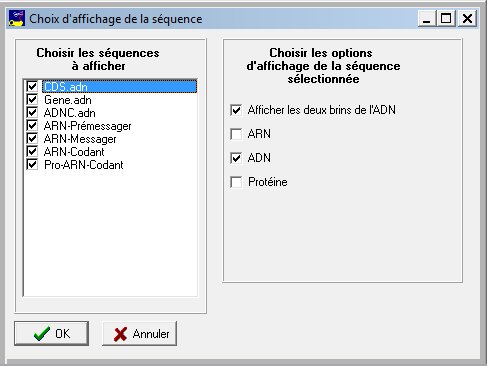 |
|
3 - Choix d'affichage par catégorie
 |
|
