Le "code barre" du vivant
Qu’est ce que le code barre du vivant (barcoding) ?
C’est une méthode qui tente de caractériser les espèces animales et végétales par le minimum de séquences d’ADN.
Pourquoi le code barre?
- Aider à la taxonomie lorsque les critères anatomo-morphologiques sont peu distinctifs des espèces.
- Mettre en évidence de nouvelles espèces dans un souci de meilleure connaissance de la biodiversité.
Quels outils pour le code barre ?

Une séquence du génome mitochondrial :
- Nombreuses copies dans une même cellule (100 à 10 000 copies).
- Le génome mitochondrial est 5 à 10 fois plus spécifique que le génome nucléaire.
- Une variabilité intra spécifique faible (perte du polymorphisme en relation avec l’hérédité maternelle du génome mitochondrial).
Ces propriétés du génome mitochondrial (faible variabilité intra spécifique et forte variabilité inter spécifique) sont à l’origine d’une véritable frontière génétique bien caractérisée entre la plupart des espèces. C’est cette propriété qui donne au génome mitochondrial la capacité d’identifier les espèces.
- Enfin, il n’y a pas d’introns dans le génome mitochondrial, ce qui rend plus facile l’amplification des séquences.
Le génome mitochondrial humain est composé de 13 gènes codant des protéines, 2 gènes codant des ARN ribosomaux et 22 gènes codant des ARN de transfert.
Le choix s’est porté sur une séquence d’environ 650 paires de bases (cela peut varier d’un phylum à l’autre) du gène codant la sous unité 1 de la cytochrome oxydase.
Une librairie de séquences suffisamment importante pour pouvoir mettre en évidence la frontière génétique intra spécifique.
Quelles sont les limites du code barre ?
- Dans les groupes où la taxonomie est bien connue (Mammifères, Oiseaux et Lépidoptères) on peut identifier un spécimen à partir d’une séquence d’ADN (expertise taxonomique). Dans les autres groupes, il reste à consolider la taxonomie avec tous les outils dont disposent les scientifiques (anatomie, morphologie, molécules…) avant de pouvoir se contenter d’un seul gène pour rattacher un spécimen à un nom d’espèce.
- Dans le cas d’espèces récemment séparées.
- Dans le cas d’insertion ponctuelle d’un fragment d’ADN dans le génome d’une autre espèce (phénomène d’introgression qui se réalise à l’occasion d’hybridations entre des individus de deux espèces différentes).
- Les pseudogènes nucléaires (copies de gènes non exprimées) peuvent également entraîner des complications dans l’identification des espèces.
L'exemple des Mammifères
Base de données : 719 spécimens.
L’analyse de la matrice de distance fait apparaître une frontière génétique nette entre les variations inter et intra spécifiques. Ce qui permet d’envisager d’utiliser la séquence CO1 dans le groupe des Mammifères pour réaliser des caractérisations d’espèces.
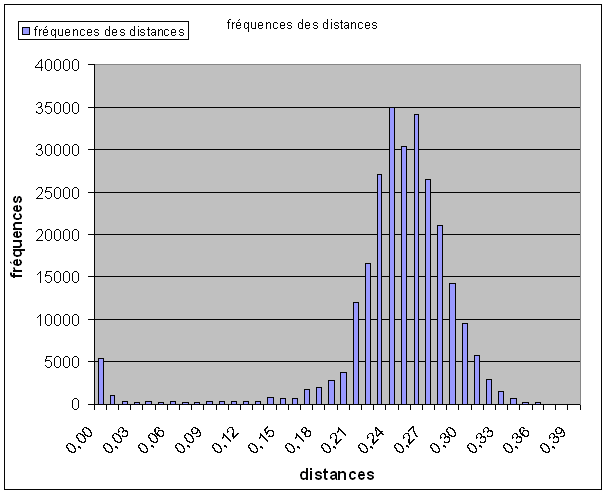
Cet histogramme correspond à deux groupes d’individus :
- Un groupe d’individus qui présentent entre leurs séquences COI des distances élevées comprises entre 0,15 et 0,36, qui correspondent à des distances entre des individus d’espèces différentes.
- Un groupe d’individus qui présentent entre leurs séquences COI des distances faibles comprises entre 0 et 0,01, qui correspondent à des distances entre des individus de mêmes espèces.
Entre ces deux « populations » il existe des distances intermédiaires mais représentant moins de 1% du total des distances mesurées.
Ces valeurs intermédiaires peuvent provenir de :
- 2 espèces taxonomiquement différentes qui n’en sont en réalité qu’une seule, ce qui se traduit par des distances associées à des variations inter-spécifiques mais qui correspondent à des variations intra-spécifiques.
- 1 espèce qui en contient en réalité 2 ce qui se traduit par des distances associées à des variations intra-spécifiques mais qui correspondent à des variations inter-spécifiques.
- Problèmes d’échantillonnage.
- Problèmes liés à la séquence COI elle-même.
Documents complémentaires
- Des diapositives pour comprendre le barcoding.
- Des séquences pour tester "un saucisson" dit "pur âne" (fichier .edi pour Anagène).
- Biodiversité, Taxonomie et Barcode moléculaire (document de Nicolas PUILLANDRE moniteur au MNHN)
